

ADMIN | 2022-02-18 07:19:16.0
感谢所有专家秉持专业和公正的态度参与本年度十大进展的推荐和评选;祝贺所有入选工作的团队!同时祝愿大家在2022新的一年里健康平安、工作顺利,如虎添翼!
评审委员会
2022年2月18日

进展题目 | 通信作者 |
宿兵,李程,张世华 | |
杜茁 | |
Chinese Glioma Genome Atlas (CGGA): a comprehensive | 江涛,保肇实 |
赵方庆 | |
| 张泽民,金荣华,陈捷凯,王晓群,瞿昆,张政,苏冰,王红阳,王福生,赵平森,李祥攀,程涛,刘新东,卞修武,魏来,贝锦新,黄志伟,蒋庆华,周鹏辉 | |
薛勇彪,鲍一明,章张,赵文明,肖景发,何顺民,张国庆,李亦学,赵国屏,陈润生 | |
黄三文 | |
张强锋 | |
谢正伟,郑瑞茂,张宁,周虹 | |
张奇伟,张新荣,陈阳 |
人类大脑起源于漫长的生命进化过程。哪些遗传改变造就了人类大脑是国际科学界长期力图回答的前沿科学问题。中国科学院昆明动物研究所宿兵团队与北京大学李程团队及中国科学院数学与系统科学研究院张世华团队合作,构建了猕猴胎脑高分辨率大脑3D基因组图谱。通过跨物种进化分析,他们发现了人类特有的染色质空间结构和脑发育调控元件,证明了人类已进化出更为精细的脑发育调控网络。该研究为阐明人类大脑特异表型的遗传机制提供了全新的线索。同时,该研究中产生的猕猴神经发育高峰期的表观多组学数据,为后续猕猴功能基因组的注释以及人类脑疾病的研究提供了极具价值的参考数据资源。
该成果发表于Cell。
数据链接:
https://ngdc.cncb.ac.cn/gsa/browse/CRA001934
原文信息:
Luo X, Liu Y, Dang D, Hu T, Hou Y, Meng X, et al. 3D genome of macaque fetal brain reveals evolutionary innovations during primate corticogenesis. Cell 2021;184:723–40. PMID: 33508230
原文链接:
https://www.sciencedirect.com/science/article/pii/S0092867421000015
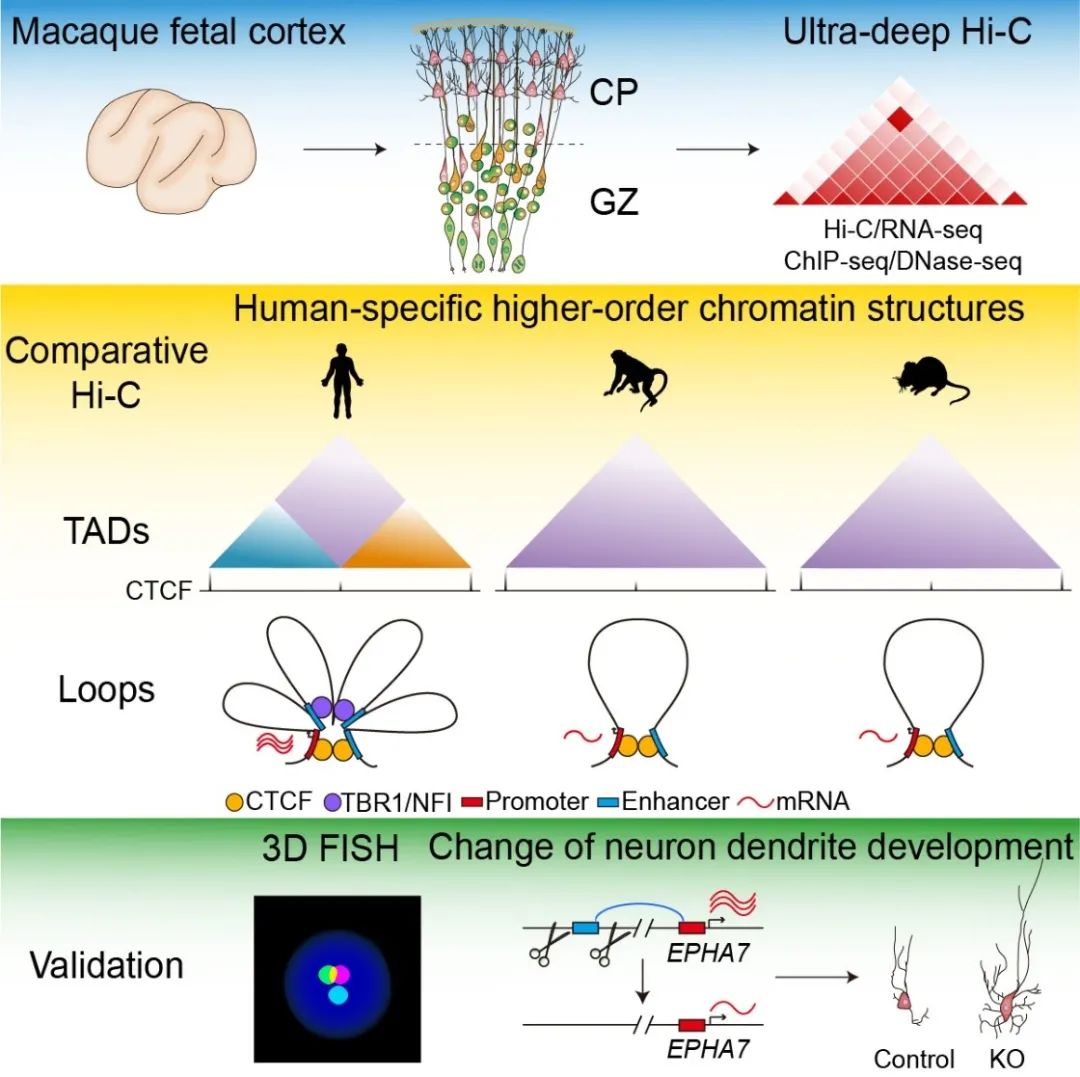
图:高分辨猕猴胎脑3D基因组图谱的构建及跨物种染色质结构进化解析发现人类特异调控元件影响神经细胞的树突发育
• 基于4D活体成像绘制秀丽线虫转录因子的单细胞空间蛋白表达图谱
阐明每个细胞的蛋白表达及功能状态是理解发育的重要切入点。中国科学院遗传与发育生物学研究所杜茁团队利用荧光报告品系,综合活体成像、实时谱系追踪和单细胞定量分析,实现了在原位以非侵入的方式解析转录因子在各个胚胎细胞的蛋白动态表达模式,并同时明确了细胞的谱系身份、空间定位及发育命运。基于图谱,研究构建了时空调控通路,揭示了转录因子新功能,归纳了细胞命运多维调控的基本框架,发现了细胞调控状态呈现高度动态性和多样性,并提出了其随发育转变的非定向“绕路”模型。该研究为理解胚胎发育提供了“高精度导航图”,推动了单细胞-全胚胎-多维度的发育定量生物学与系统生物学研究。
该成果发表于Nature Methods。
数据库链接:
http://dulab.genetics.ac.cn/TF-atlas/
原文信息:
Ma X, Zhao Z, Xiao L, Xu W, Kou Y, Zhang Y, et al. A 4D single-cell protein atlas of transcription factors delineates spatiotemporal patterning during embryogenesis. Nat Methods 2021;18:893–902. PMID: 34312566
原文链接:
https://www.nature.com/articles/s41592-021-01216-1
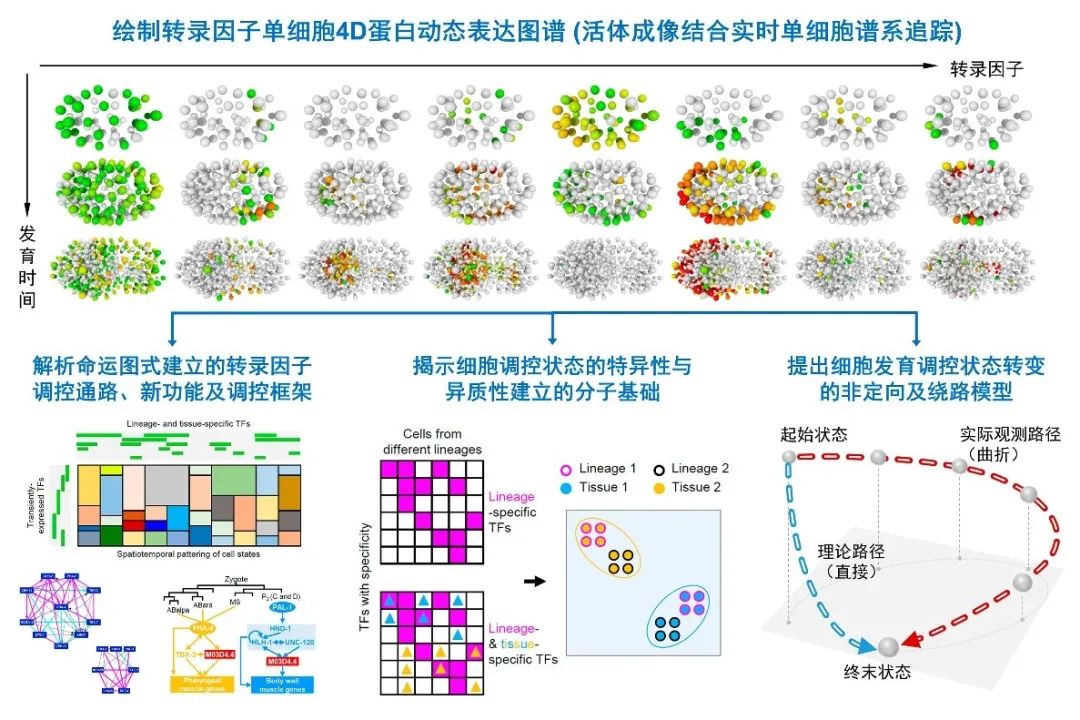
图:转录因子单细胞蛋白动态表达图谱揭示发育图式建立的分子调控基本框架
• 中国脑胶质瘤基因组图谱计划(Chinese Glioma Genome Atlas,CGGA)数据库
脑胶质瘤作为成人最常见的、极难治疗的颅内恶性肿瘤,其基于多维组学的遗传特征和生物学功能亟待挖掘和研究。北京市神经外科研究所江涛/保肇实团队,针对中国人群,构建了大规模多中心中国脑胶质瘤基因组图谱计划数据库(CGGA)。该数据库拥有全球范围内覆盖全面病理亚型的脑胶质瘤样本,包括低级别、高级别、复发脑胶质瘤等。最长随访时间逾15年。其中2000余例样本的多维组学数据已实现云储存和共享下载,包括全外显子组、转录组、表观遗传组学数据、单细胞数据等。CGGA网站还可实现数据的在线分析,并提供用户友好的可视化分析工具。该数据库为目前国际上脑胶质瘤病理类型最全面、种类独特的脑胶质瘤临床样本最多的多维组学数据库。
该成果发表于Genomics, Proteomics & Bioinformatics。
数据库链接:
原文信息:
Zhao Z, Zhang KN, Wang Q, Li G, Zeng F, Zhang Y, et al. Chinese Glioma Genome Atlas (CGGA): a comprehensive resource with functional genomic data from Chinese glioma patients. Genomics Proteomics Bioinformatics 2021;19:1–12. PMID: 533662628
原文链接:
https://www.sciencedirect.com/science/article/pii/S1672022921000450?via%3Dihub
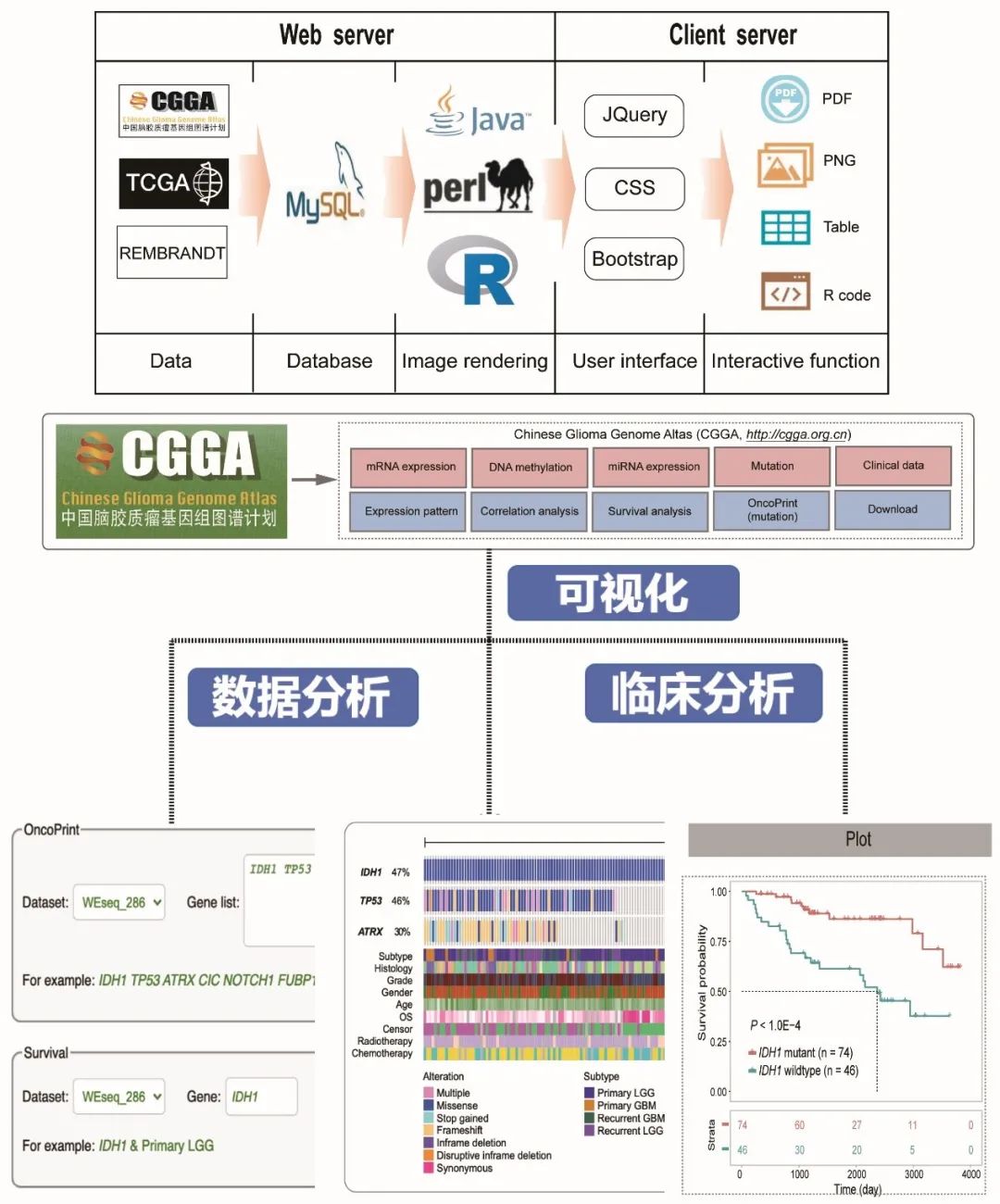
图:CGGA数据库开发结构示意图及功能实现
环形RNA是一类在真核生物中广泛存在的环状转录本,在生物体内通过其序列特征发挥微小RNA海绵、RNA结合蛋白(RBP)海绵及翻译小肽等重要的生物学功能。因此,确定环形RNA的全长序列,是研究环形RNA功能的重要基础。中国科学院北京生命科学研究院赵方庆团队通过结合滚环反转录扩增和三代纳米孔测序技术,开发了高效测定环形RNA全长转录本的实验与计算方法CIRI-long,解决了目前研究方法中难以区分环形RNA与线性mRNA来源读段的问题,实现了不同长度环形RNA的高灵敏度检测和内部结构重构。利用该方法,研究团队鉴定到了大量环形RNA内部的可变剪接事件,并对来自线粒体基因组、相邻基因转录通读以及内含子自连产生的环形RNA分子进行了全面识别。该方法实现了环形RNA的高效识别与全长重构,为挖掘具有生物学功能的环形RNA提供了重要的研究工具。
该成果发表于Nature Biotechnology。
工具链接:
https://github.com/bioinfo-biols/CIRI-long
原文信息:
Zhang J, Hou L, Zuo Z, Ji P, Zhang X, Xue Y, et al. Comprehensive profiling of circular RNAs with nanopore sequencing and CIRI-long. Nat Biotechnol 2021;39:836–45. PMID: 33707777
原文链接:
https://www.nature.com/articles/s41587-021-00842-6
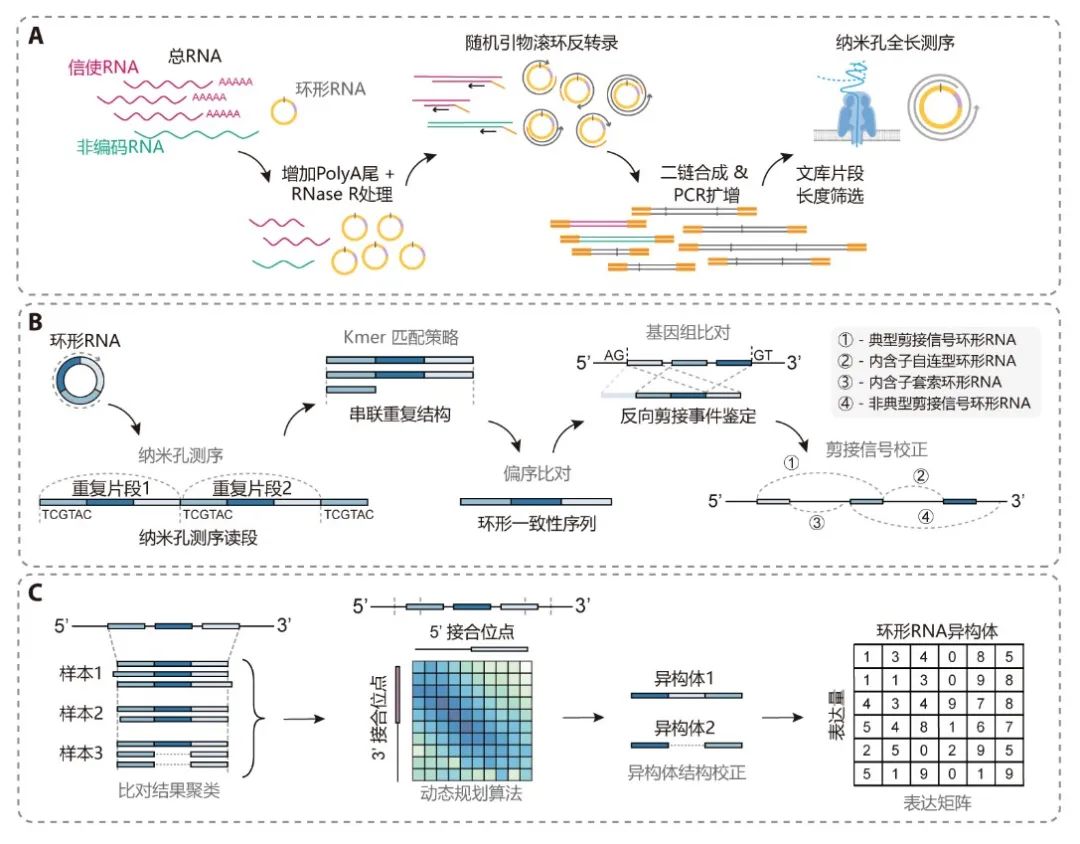
图:环形RNA的准确识别及全长重构
环形RNA的实验富集(A),全长识别(B),类型注释及多样本数据整合分析(C)。
新冠(COVID-19)疫情爆发伊始,北京大学、首都医科大学附属北京佑安医院、中国科学院生物物理研究所、中国科学技术大学、深圳第三人民医院、上海交通大学、海军军医大学、中国人民解放军总医院第五医学中心、粤北人民医院、武汉大学人民医院、中国医学科学院血液病医院(血液学研究所)、陆军军医大学、哈尔滨工业大学、中山大学、广州生物岛实验室、深圳湾实验室等40多家科研单位迅速组建了“新冠单细胞研究中国联盟”,对轻症、重症COVID-19病人包括健康对照的外周血、肺泡灌洗液等不同类型样本进行了单细胞转录组测序和生物信息分析。研究发现,新冠病毒核酸在上皮细胞与巨噬细胞、中性粒细胞、T细胞等多种免疫细胞中均可检出,且具有亚基因组转录的特点,提示新冠病毒在人体中具有广泛的宿主细胞谱。配体-受体分析显示,感染新冠病毒后纤毛上皮细胞倾向于脱落,而鳞状样上皮细胞会上调S100A8/9、ANXA1等因子的基因表达,通过与TLR4、FPR1相互作用过度招募巨噬细胞与中性粒细胞从而导致肺炎。本研究还揭示COVID-19病人外周血中存在一群高表达S100A8/9、CCL3等细胞因子基因的单核细胞,是导致细胞因子风暴的重要源头。这些发现对认识和控制新冠疫情具有重要指导意义。
该成果发表于Cell。
数据链接:
https://ngdc.cncb.ac.cn/gsa-human/browse/HRA001149
原文信息:
Ren X, Wen W, Fan X, Hou W, Su B, Cai P, et al. COVID-19 immune features revealed by a large-scale single-cell transcriptome atlas. Cell 2021;184:1895–1913.e19. PMID: 33657410
原文链接:
https://www.sciencedirect.com/science/article/pii/S0092867421001483?via%3Dihub
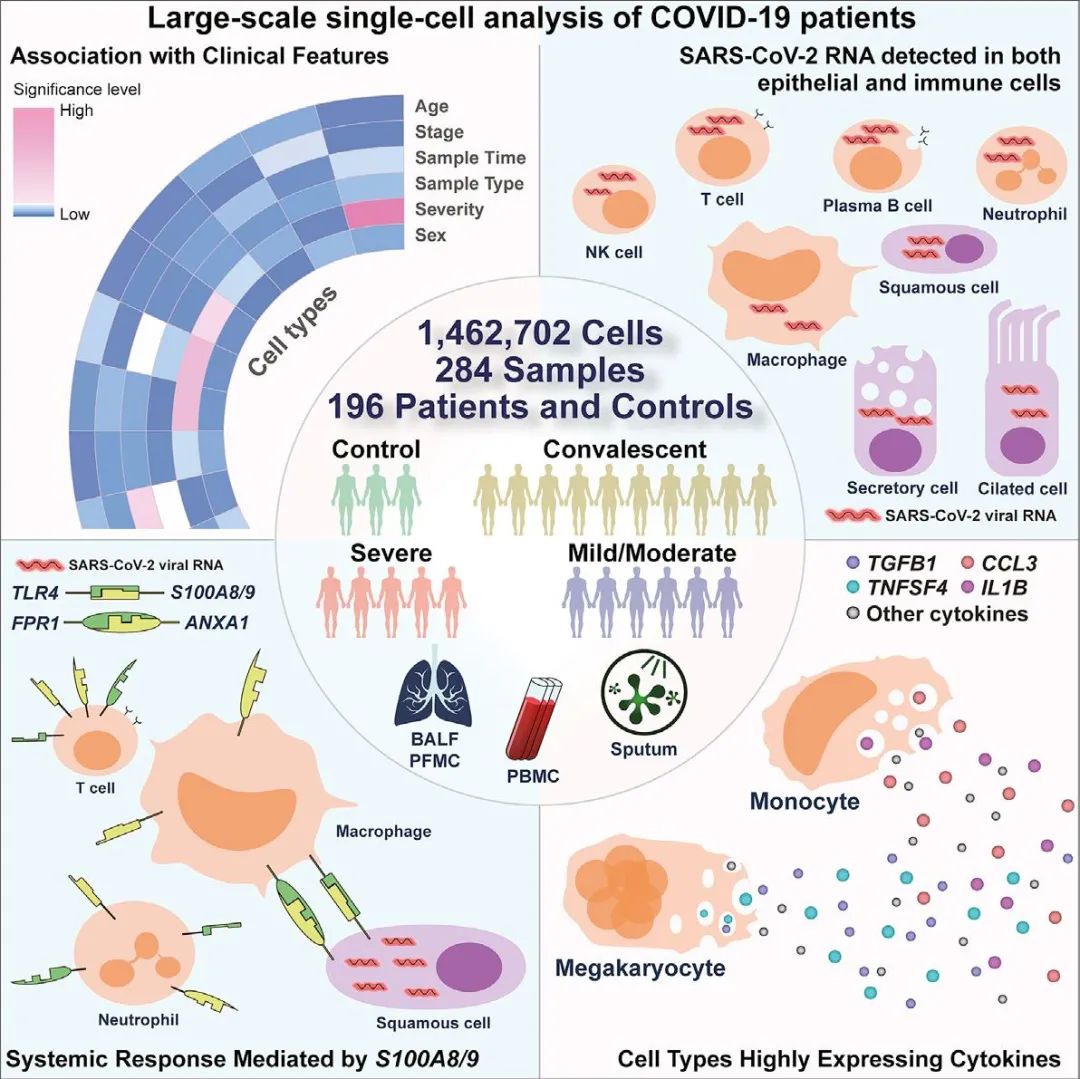
图:单细胞转录组测序揭示新冠肺炎重要感染与免疫机制
中心:实验设计与样本类型;左上:外周血中不同免疫细胞与疾病严重程度、年龄、性别等存在不同关联;右上:上皮细胞与免疫细胞中可检出新冠病毒核酸;左下:鳞状样上皮细胞在招募巨噬细胞中性粒细胞中的关键作用;右下:细胞因子风暴的潜在来源。
基因组科学数据是人口健康和国家安全的重要战略资源。为存好、管好和用好基因组科学数据,中国科学院北京基因组研究所(国家生物信息中心)国家基因组科学数据中心(China National Center for Bioinformation -the National Genomics Data Center,CNCB-NGDC)面向国家大数据和健康中国战略,建成涵盖国家人类遗传资源和重要战略生物资源的多组学数据资源体系,解决了长期以来我国基因组科学数据汇交共享严重依赖国际数据库的问题,为国家基因组科学数据的汇交共享、安全管理和挖掘利用提供重要支撑。
该成果发表于Nucleic Acids Research。
数据库链接:
原文信息:
CNCB-NGDC Members and Partners. Database resources of the National Genomics Data Center, China National Center for Bioinformation in 2021. Nucleic Acids Res 2021;49:D18–8. PMID: 33175170
原文链接:
https://academic.oup.com/nar/article/49/D1/D18/5974090
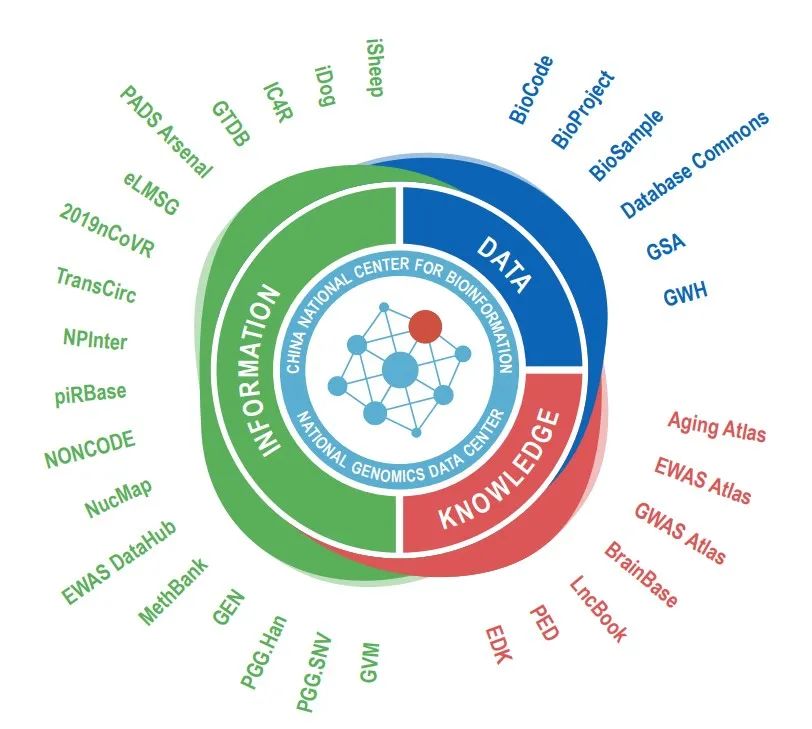
图:国家生物信息中心-国家基因组科学数据中心数据资源(2021)
为了解决马铃薯产业面临的育种周期长、薯块繁殖成本高的问题,在前期克服了自交不亲和和解析自交衰退的遗传机制的基础上,中国农业科学院农业基因组研究所黄三文研究团队联合云南师范大学等国内外优势单位,运用“基因组设计”的理论和方法体系培育杂交马铃薯,用二倍体育种替代四倍体育种,最终获得优良的杂交种子,颠覆了传统的薯块繁殖方式。该研究培育出第一代高纯合度(> 99%)自交系,以及具有显著的产量杂种优势的杂交品系“优薯1号”,证明了杂交马铃薯育种的可行性,使马铃薯的遗传改良取得了里程碑式突破。
该成果发表于Cell。
数据链接:
https://www.ncbi.nlm.nih.gov/bioproject/PRJNA641265/;
https://bigd.big.ac.cn/gvm/getProjectDetail?Project=GVM000101;
https://github.com/DieTANG/PiGBS_Pipeline
原文信息:
Zhang C, Yang Z, Tang D, Zhu Y, Wang P, Li D, et al. Genome design of hybrid potato. Cell 2021;184:3873–83.e12. PMID: 34171306
原文链接:
https://www.sciencedirect.com/science/article/pii/S0092867421007078?via%3Dihub
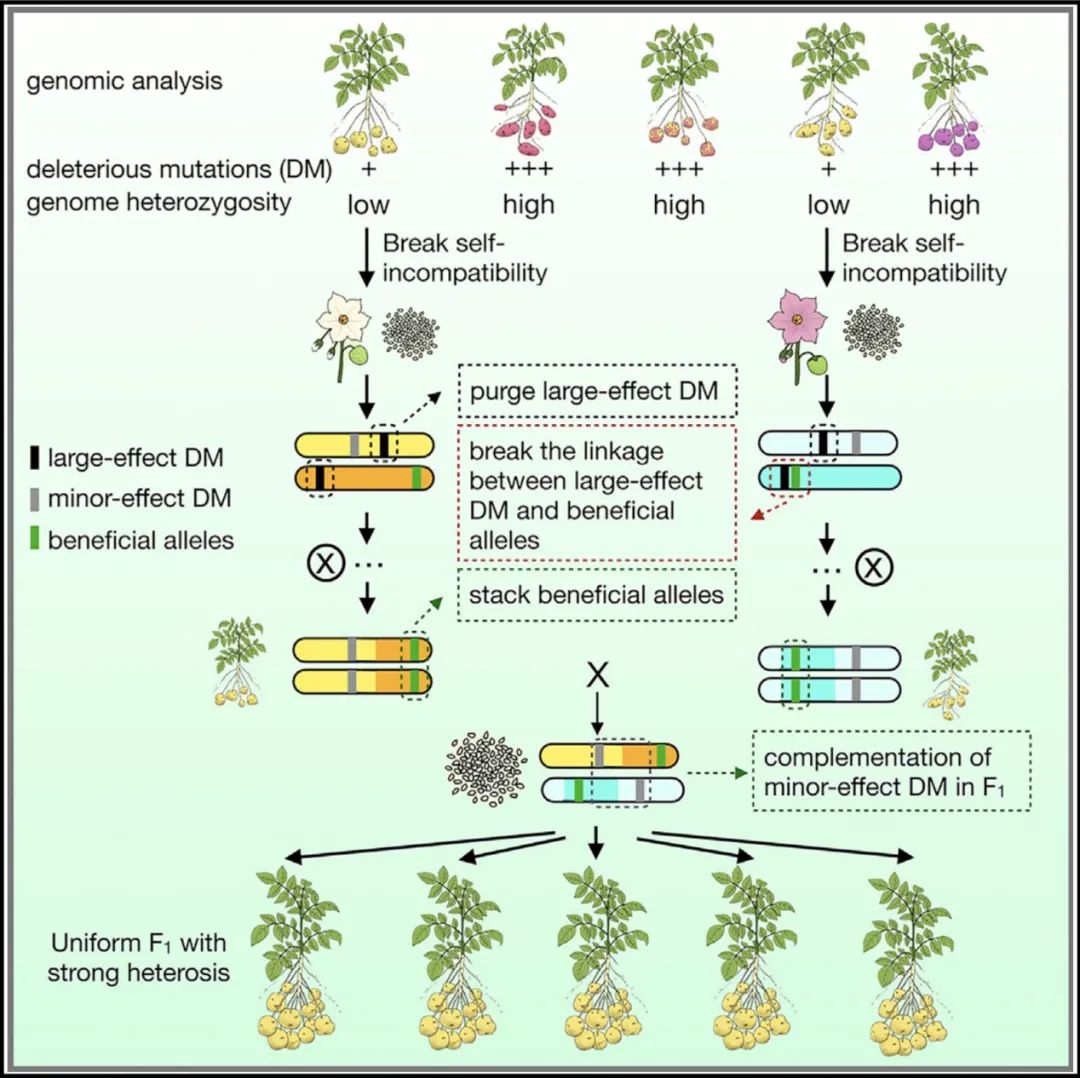
图: 杂交马铃薯的基因组设计原理图
RBP在RNA分子的整个生命周期包括转录、代谢、翻译以及降解等过程中动态结合RNA分子,对RNA分子的命运起着关键调控作用。清华大学张强锋团队基于细胞内RNA结构与对应细胞环境的RBP结合信息的耦合关系,建立了利用细胞内RNA结构信息预测细胞内RBP动态结合的人工智能新方法PrismNet。对于任意一个RBP,只要在某一种细胞环境下做了crosslinking and immunprecipitation(CLIP)实验,PrismNet就可以通过构建准确的深度神经网络模型,把结合信息外推到其他细胞环境中。在PrismNet的一个应用研究中,利用新冠病毒在宿主细胞内的RNA结构信息,研究组准确预测了多个新冠病毒的宿主结合蛋白,证明了PrismNet的广阔应用前景。
该成果发表于Cell Research。
工具链接:
http://prismnetweb.zhanglab.net/
原文信息:
Sun L, Xu K, Huang W, Yang YT, Li P, Tang L, et al. Predicting dynamic cellular protein–RNA interactions by deep learning using in vivo RNA structures. Cell Res 2021;31:495–516. PMID: 33623109
原文链接:
https://www.nature.com/articles/s41422-021-00476-y
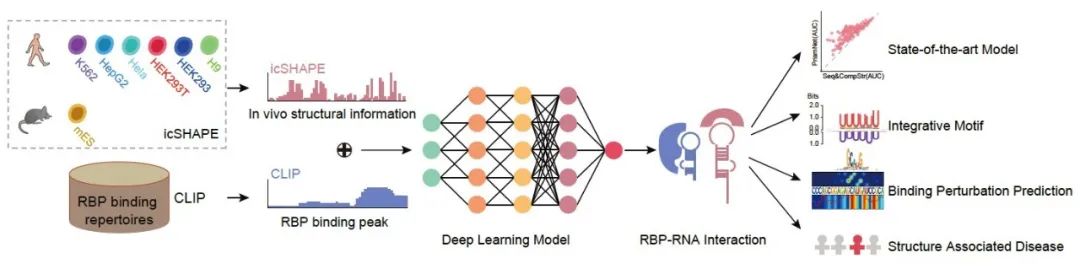
图:PrismNet模型构建以及应用
创新药物研发是一个周期长、耗费大的艰难过程。在药物研发方面,人工智能/深度学习(AI制药)被寄予厚望。北京大学谢正伟、郑瑞茂、张宁和周虹联合研究团队首先构建了一个神经网络,使用简化分子线性输入规范(SMILES)化学编码作为输入,以预测L1000数据中测量的转录组变化。然后使用疾病相关基因指纹来反映特定疾病的“内在痕迹”,通过基因集合富集分析(GSEA)来评估化合物对疾病的潜在疗效。研究人员将这种方法和模型称为基于深度学习的药效预测系统(DLEPS; 中文名为灵素系统)。研究员人员针对商用小分子数据库预测了治疗三种常见慢性病的化合物,并进行了实验验证,在每种疾病研究方向都发现了疗效优越的小分子。该系统是国际上第一款药效预测系统,受到国际同行的高度评价和广泛关注。
该成果发表于Nature Biotechnology。
工具链接:
https://www.dleps.tech/dleps/index;
https://github.com/kekegg/DLEPS
原文信息:
Zhu J, Wang J, Wang X, Gao M, Guo B, Gao M, et al. Prediction of drug efficacy from transcriptional profiles with deep learning. Nat Biotechnol 2021;39:1444–52. PMID: 34140681
原文链接:
https://www.nature.com/articles/s41587-021-00946-z
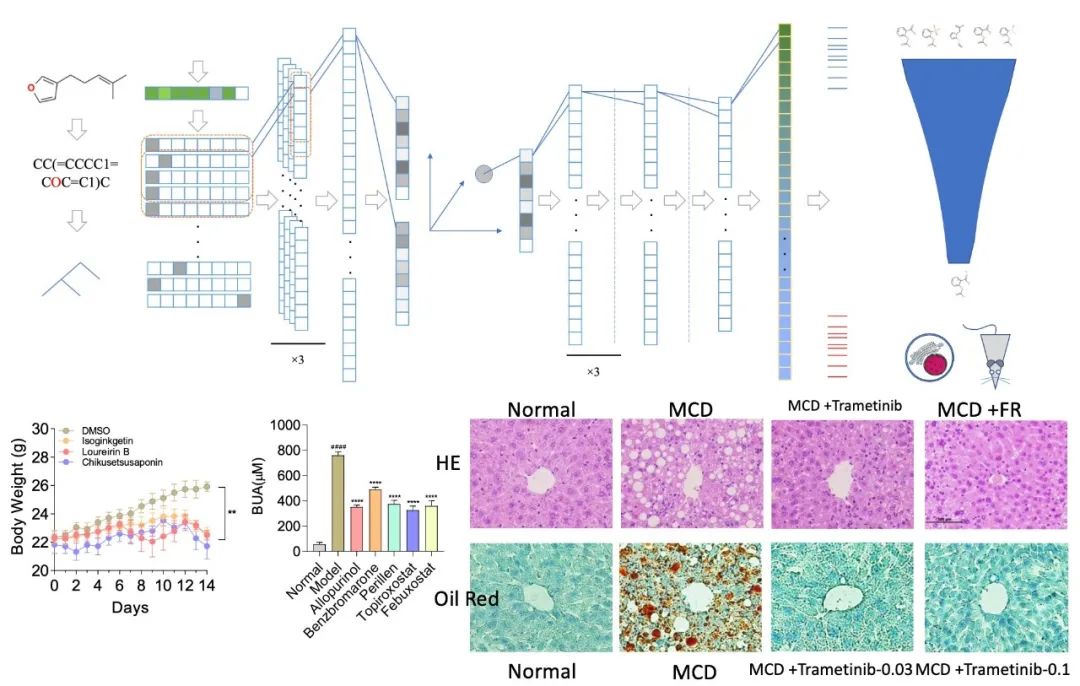
图:深度神经网络的结构和原理图以及DLEPS在肥胖、尿酸血症和非酒精性脂肪性肝炎中的应用效果
机体中的代谢分子可以显著影响基因表达,最终改变细胞命运。因此,在组织原位观测细胞核中的代谢组特征对认识机体发育、肿瘤发生发展、神经系统衰老等生命过程至关重要。结合高空间分辨率成像质谱技术和人工智能算法,清华大学张奇伟教授、张新荣教授、陈阳副研究员(现北京协和医学院研究员)带领跨学科交叉合作团队在国际上首次建立了空间单细胞核代谢组技术SEAM。该技术可定位单个细胞在组织网络中的位置、区分每个细胞相关代谢物的指纹图谱差异、确定重要代谢物的分子组成,推动“空间代谢组技术进入了亚细胞时代”。
该成果发表于Nature Methods。
方法链接:
https://github.com/yuanzhiyuan/SEAM/
原文信息:
Yuan Z, Zhou Q, Cai L, Pan L, Sun W, Qumu S, et al. SEAM is a spatial single nuclear metabolomics method for dissecting tissue microenvironment. Nat Methods 2021;18:1223–32. PMID:34608315
原文链接:
https://www.nature.com/articles/s41592-021-01276-3
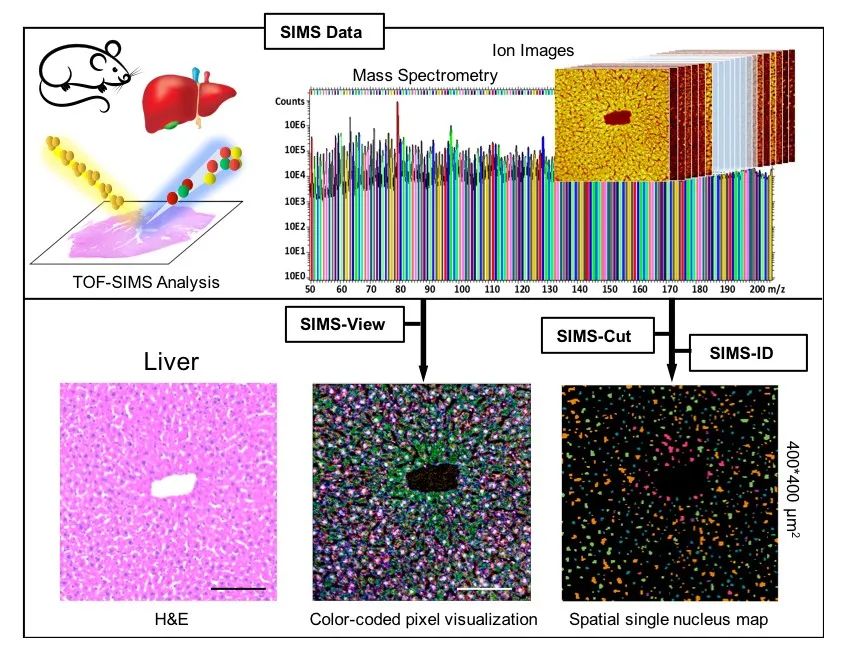
图:首个空间单细胞核代谢组分析技术SEAM