

ADMIN | 2024-03-16 12:50:41.0

为推动我国生物信息学的学科发展和创新研究,充分展示和宣传我国生物信息学领域的重大研究成果,《基因组蛋白质组与生物信息学报(英文)》(Genomics, Proteomics & Bioinformatics, 简称GPB)组织评选了2018年度、2019年度、2020年度、2021年度和2022年度“中国生物信息学十大进展”。在此基础上,GPB继续组织2023年度评选活动,经过100余名国内外生物信息学领域教授/研究员推荐,初选、复选投票,以及复核程序,现公布2023年度“中国生物信息学十大进展”评选结果(排名不分先后,按标题首字母顺序排序)。
感谢所有专家秉持专业和公正的态度参与本年度十大进展的推荐和评选;祝贺所有入选工作的团队!同时祝愿大家在2024新的一年里健康平安、工作顺利、大展宏图、硕果累累!
评审委员会
2024年3月16日

泛癌种自然杀伤细胞异质性的生物信息学解析
该成果发表于Cell
推荐理由:首个人类自然杀伤细胞的泛癌图谱
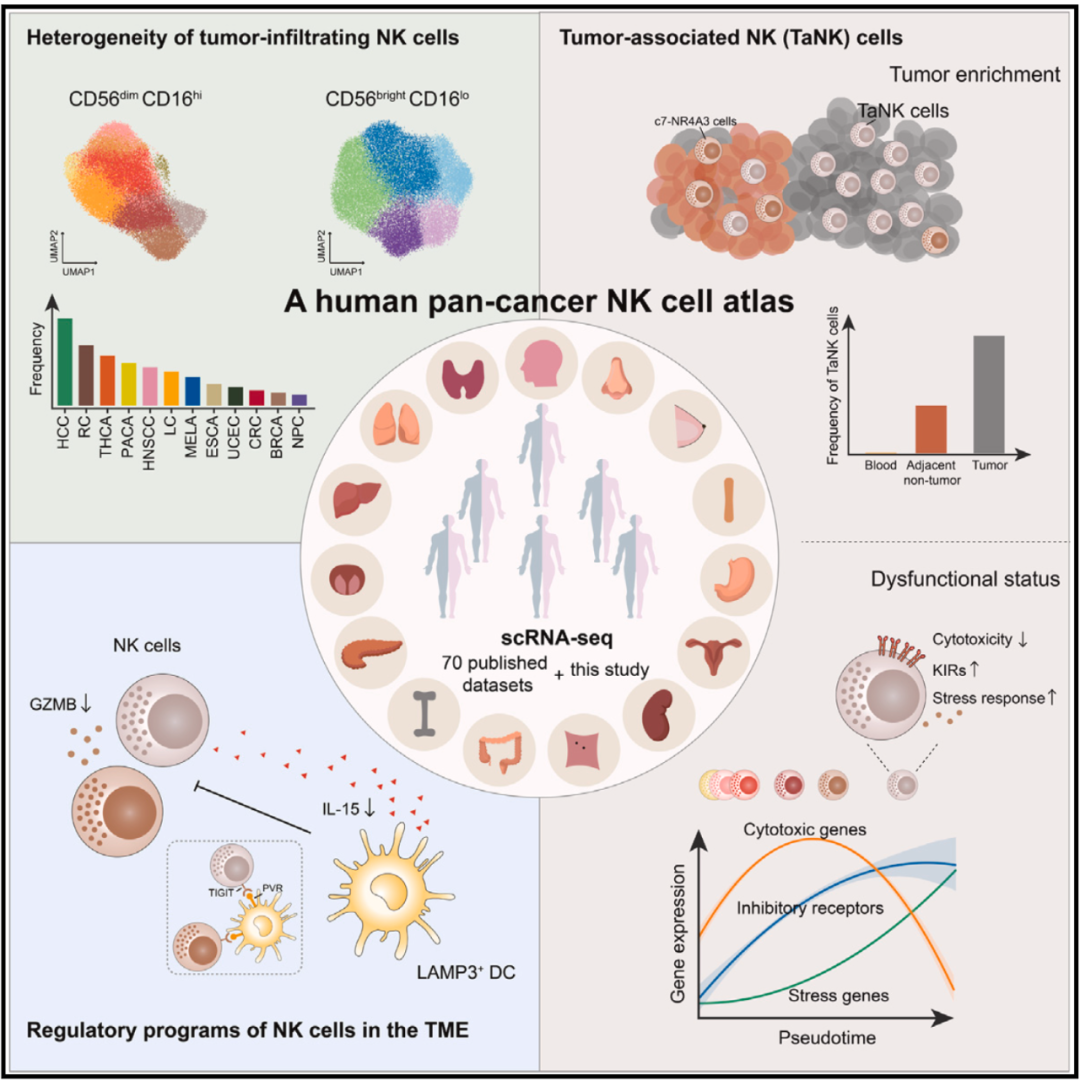
图:生物信息学整合分析揭示NK细胞的泛癌种异质性规律
数据链接
http://pan-nk.cancer-pku.cn/
原文信息
Tang F, Li J, Qi L, Liu D, Bo Y, Qin S, et al. A pan-cancer single-cell panorama of human natural killer cells. Cell 2023;186:4235–51.e20. PMID: 37607536.
原文链接
https://doi.org/10.1016/j.cell.2023.07.034

▲ 长按阅读原文
人类和小鼠细胞身份识别及单细胞功能分析平台——CellMarker 2.0
该成果发表于Nucleic Acids Research
推荐理由:提供了人/鼠不同细胞类型的分子标志物的高质量数据
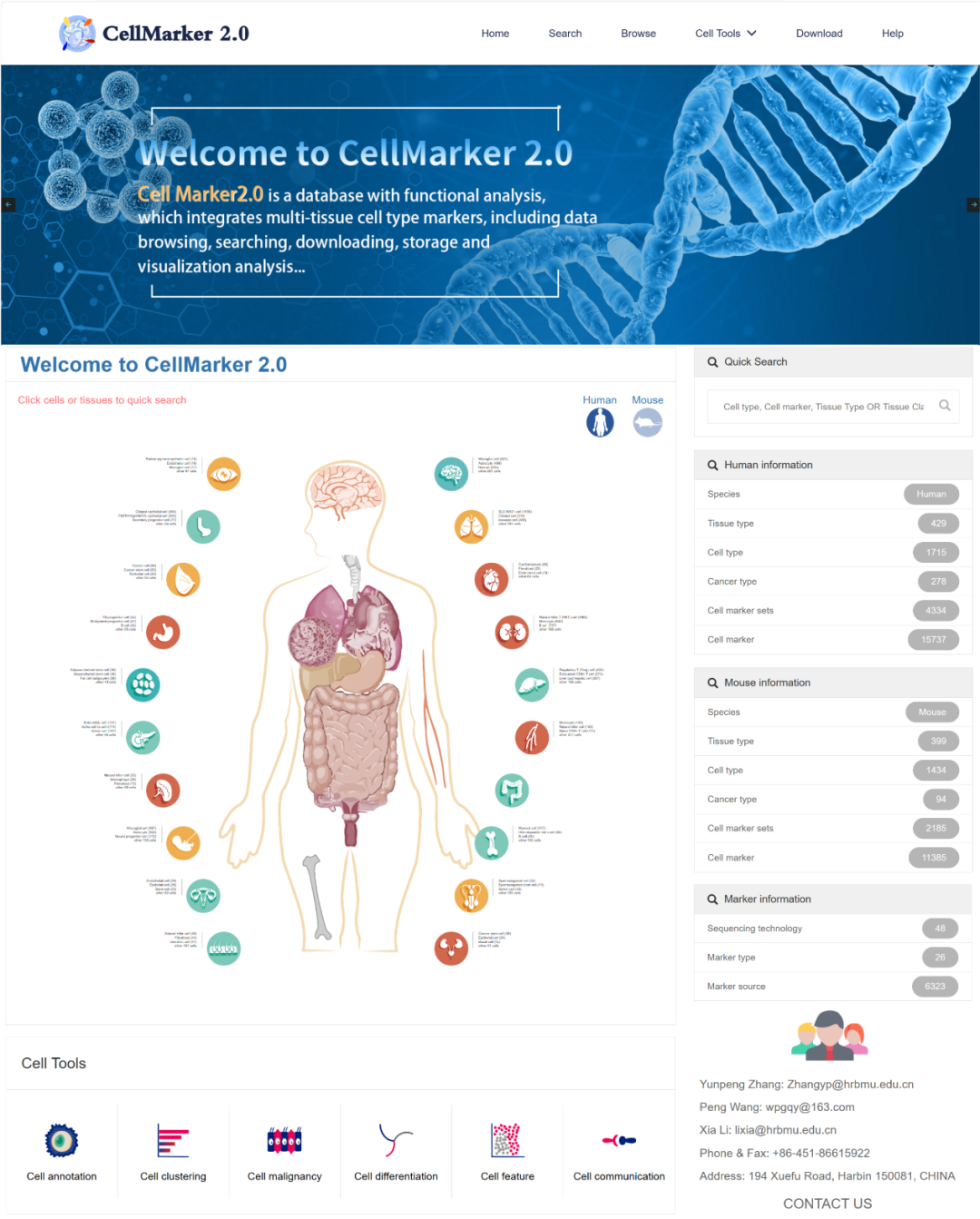
图:CellMarker 2.0细胞身份识别及单细胞功能分析平台
数据库链接
http://bio-bigdata.hrbmu.edu.cn/CellMarker/
http://117.50.127.228/CellMarker/
原文信息
Hu C, Li T, Xu Y, Zhang X, Li F, Bai J, et al. CellMarker 2.0: an updated database of manually curated cell markers in human/mouse and web tools based on scRNA-seq data. Nucleic Acids Research 2023;51:D870–6. PMID: 36300619.
原文链接
https://academic.oup.com/nar/article/51/D1/D870/6775381

▲ 长按阅读原文
揭示基因组重复序列Alu调控转录新机制
该成果发表于Nature
推荐理由:揭示了增强子与启动子交互的选择性
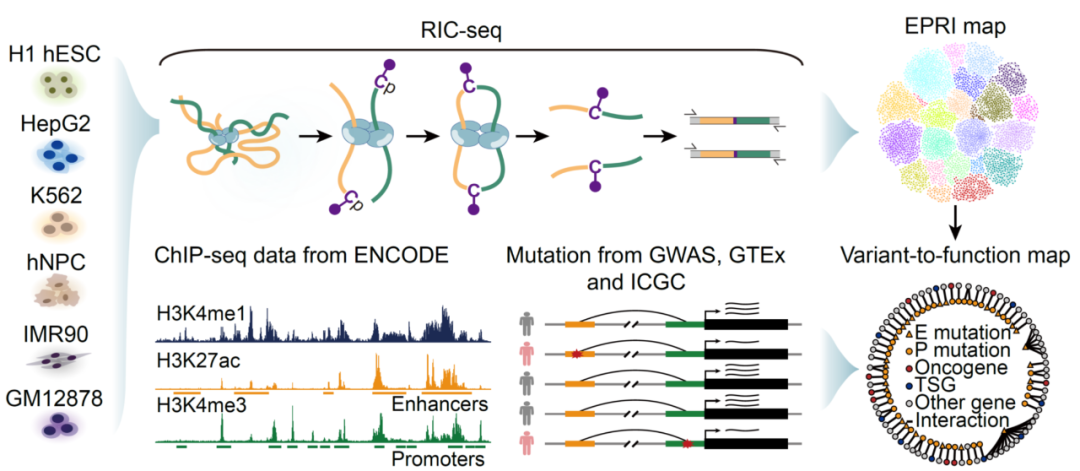
图:“增强子-启动子互作图谱”以及“突变-功能图谱”构建
原文信息
Liang L, Cao C, Ji L, Cai Z, Wang D, Ye R, et al. Complementary Alu sequences mediate enhancer-promoter selectivity. Nature 2023;619:868–75. PMID: 37438529.
原文链接
https://doi.org/10.1038/s41586-023-06323-x

▲ 长按阅读原文
我国生命组学数据资源体系建设成效显著
该成果发表于Nucleic Acids Research
推荐理由:筑造中国生物数据资源根基
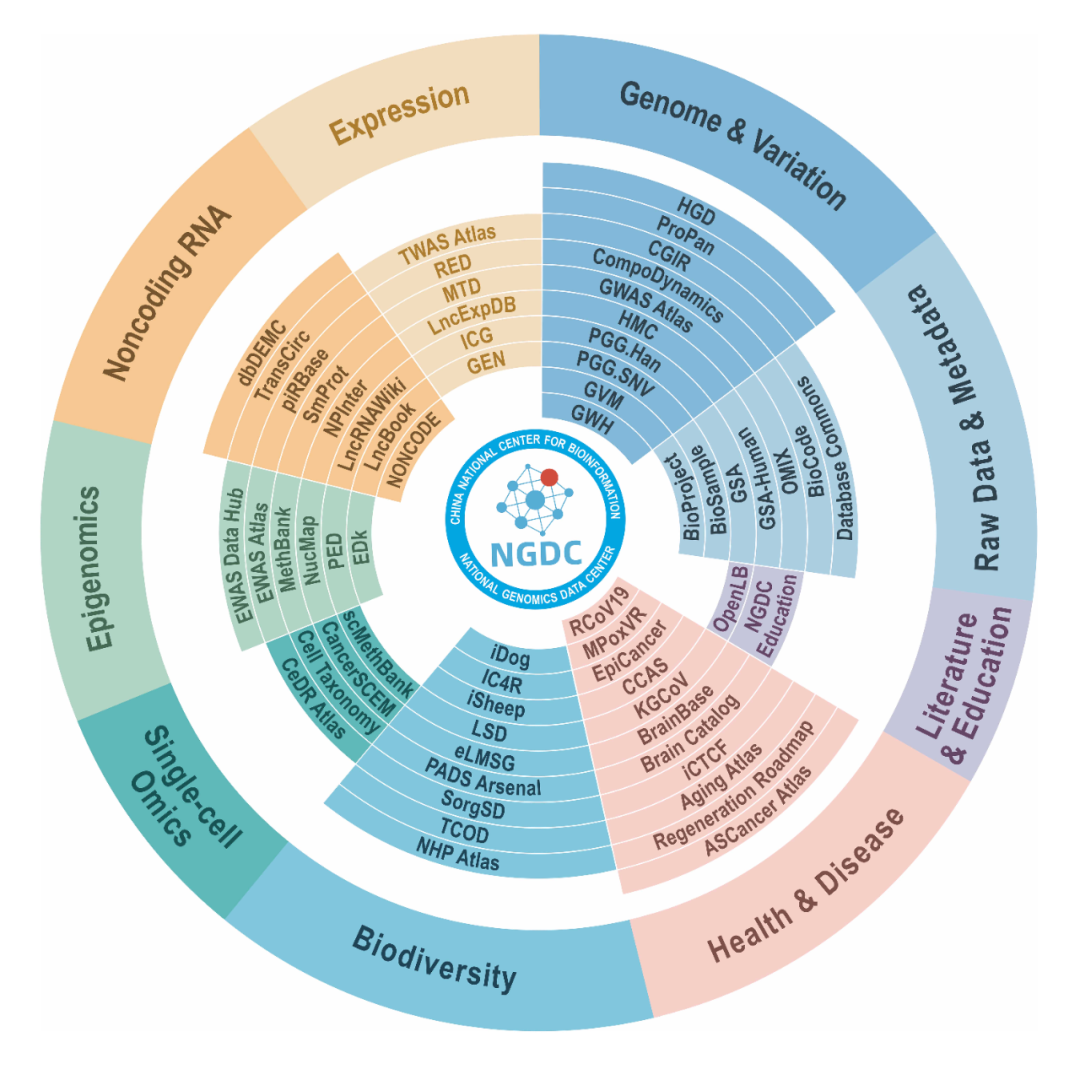
图:CNCB-NGDC多组学数据资源体系
数据库链接
https://ngdc.cncb.ac.cn
原文信息
CNCB-NGDC Members and Partners. Database resources of the National Genomics Data Center, China National Center for Bioinformation in 2023. Nucleic Acids Research 2023;51:D18–28. PMID: 36420893.
原文链接
https://academic.oup.com/nar/article/51/D1/D18/6845434

▲ 长按阅读原文
结构驱动的碱基编辑器开发与应用
该成果发表于Cell
推荐理由:人工智能指导的蛋白质结构聚类,助力研发新型碱基编辑工具
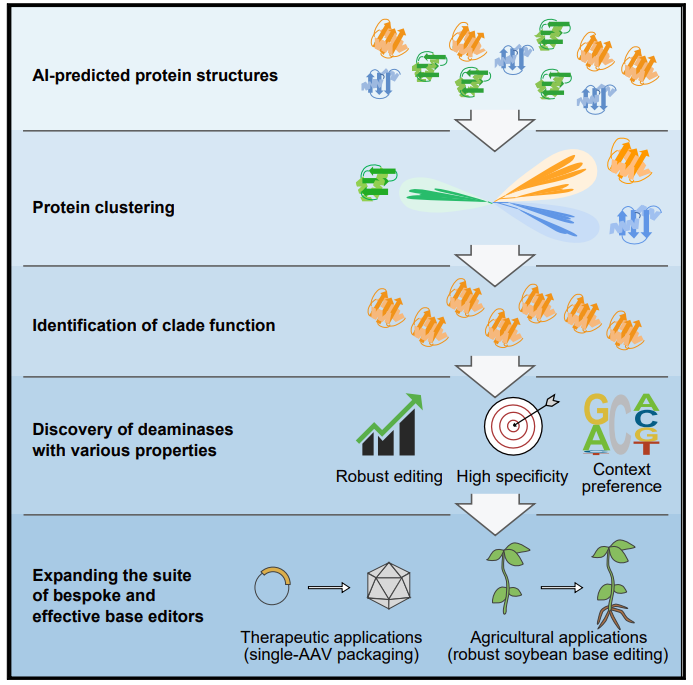
图:新型碱基编辑器开发与应用
数据链接
https://www.ncbi.nlm.nih.gov/bioproject/PRJNA915939/
https://www.ncbi.nlm.nih.gov/bioproject/PRJNA915940/
https://www.ncbi.nlm.nih.gov/bioproject/PRJNA915941/
https://www.ncbi.nlm.nih.gov/bioproject/PRJNA915942/
https://www.addgene.org/browse/article/28238292/
原文信息
Huang J, Lin Q, Fei H, He Z, Xu H, Li Y, et al. Discovery of deaminase functions by structure-based protein clustering. Cell 2023;186:3182–95.e14. PMID: 37379837.
原文链接
https://www.cell.com/cell/abstract/S0092-8674(23)00593-7

▲ 长按阅读原文
新方法实现单细胞命运轨迹的精确预测——PhyloVelo
该成果发表于Nature Biotechnology
推荐理由:基于谱系示踪信息精确计算RNA速率的新方法
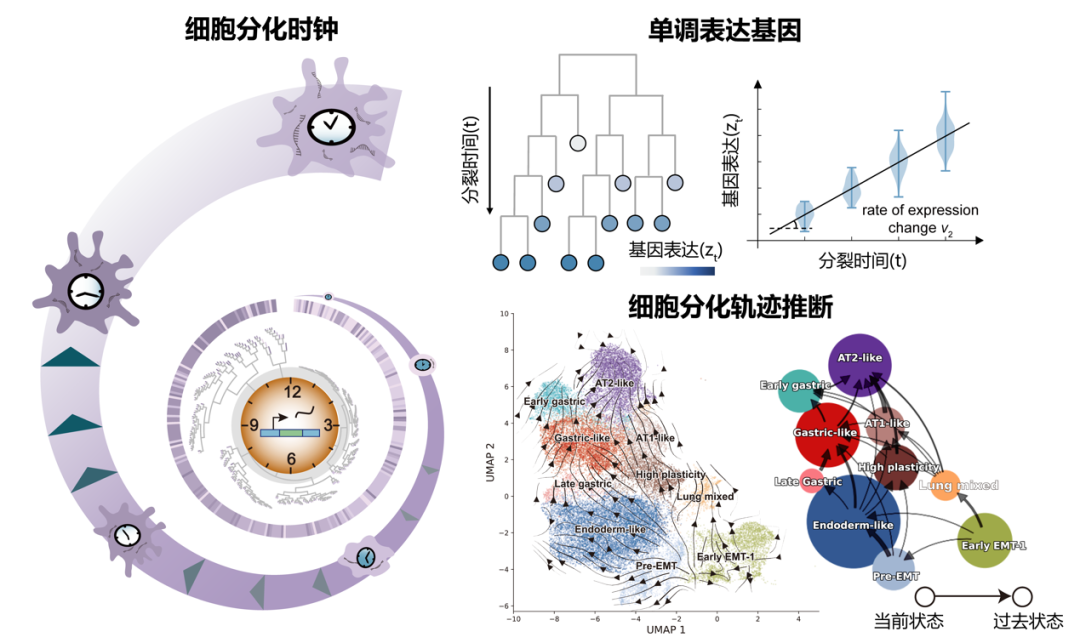
图:基于单调表达基因的细胞分化轨迹推断新框架(PhyloVelo)
在线使用文档
https://phylovelo.readthedocs.io/en/latest/index.html
原文信息
Wang K, Hou L, Wang X, Zhai X, Lu Z, Zi Z, et al. PhyloVelo enhances transcriptomic velocity field mapping using monotonically expressed genes. Nature Biotechnology 2023. https://doi.org/10.1038/s41587-023-01887-5. PMID: 37524958.
原文链接
https://www.nature.com/articles/s41587-023-01887-5

▲ 长按阅读原文
单液滴细胞外囊泡异质性解析新技术——SEVtras
该成果发表于Nature Methods
推荐理由:从单细胞转录组数据中解码胞外小囊泡的异质性
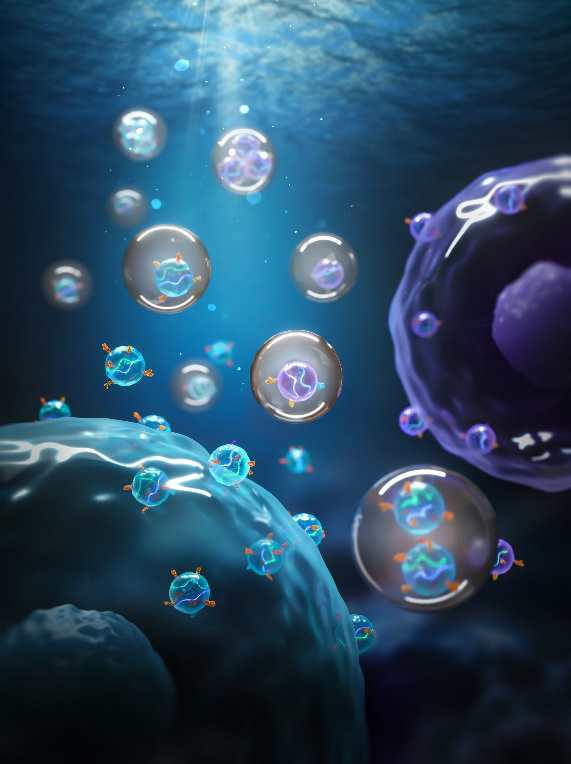
图:SEVtras追踪胞外小囊泡异质性
工具链接
https://github.com/bioinfo-biols/SEVtras
原文信息
He R, Zhu J, Ji P, Zhao F. SEVtras delineates small extracellular vesicles at droplet resolution from single-cell transcriptomes. Nature Methods 2024;21:259–66. PMID: 38049696.
原文链接
https://doi.org/10.1038/s41592-023-02117-1

▲ 长按阅读原文
空间多组学数据库及分析算法——SODB
该成果发表于Nature Methods
推荐理由:领域最大的空间多组学数据库
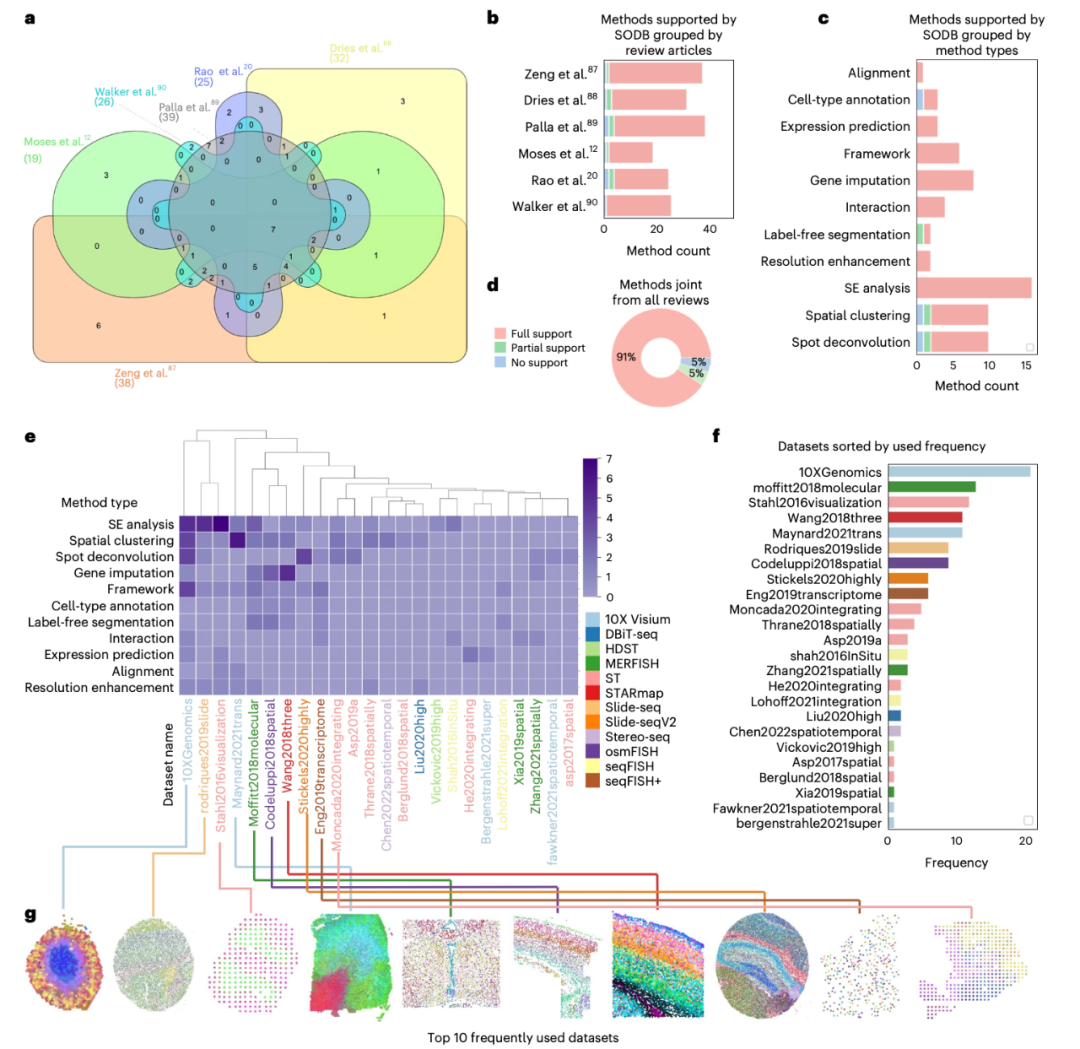
图:SODB支持多种算法的基准研究
数据库链接
https://gene.ai.tencent.com/SpatialOmics/
原文信息
Yuan Z, Pan W, Zhao X, Zhao F, Xu Z, Li X. et al. SODB facilitates comprehensive exploration of spatial omics data. Nature Methods 2023;20:387–99. PMID: 36797409.
原文链接
https://doi.org/10.1038/s41592-023-01773-7

▲ 长按阅读原文
肿瘤免疫治疗相关的基因表达资源——TIGER
该成果发表于Genomics, Proteomics & Bioinformatics
推荐理由:肿瘤免疫治疗分子标志物分析的大数据平台
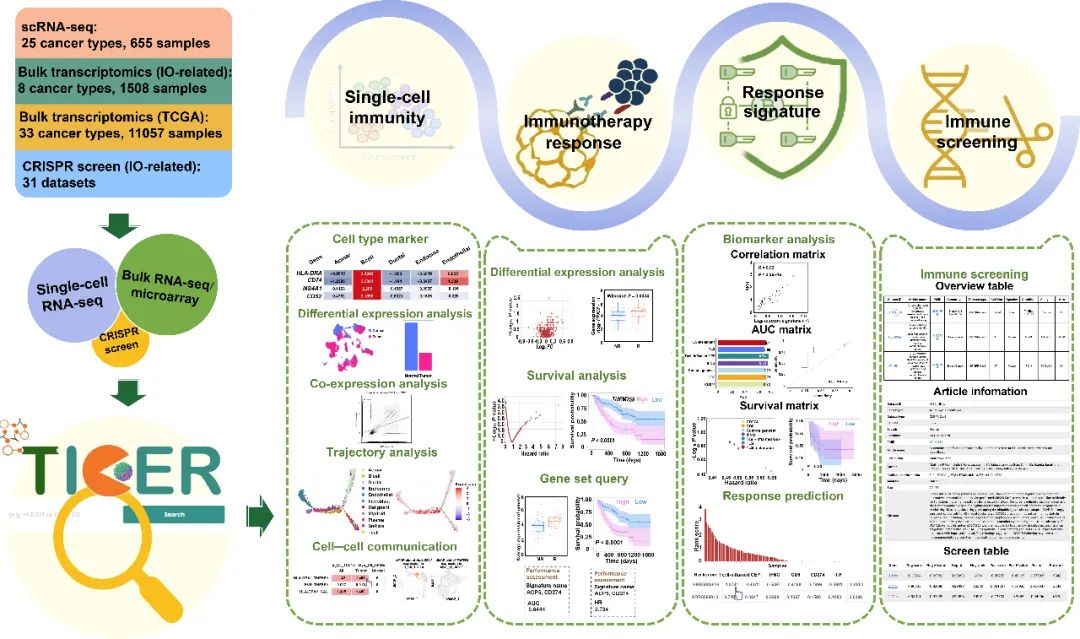
图:TIGER整体设计概览
数据库链接
http://tiger.canceromics.org/
原文信息
Chen Z, Luo Z, Zhang D, Li H, Liu X, Zhu K, et al. TIGER: a web portal of Tumor Immunotherapy Gene Expression Resource. Genomics, Proteomics & Bioinformatics 2023;21:337–48. PMID: 36049666.
原文链接
https://doi.org/10.1016/j.gpb.2022.08.004

▲ 长按阅读原文
基于工程化纳米孔的氨基酸及其翻译后修饰检测
该成果发表于Nature Methods
推荐理由:首个能够识别20种天然蛋白质氨基酸的纳米孔测序和分析技术
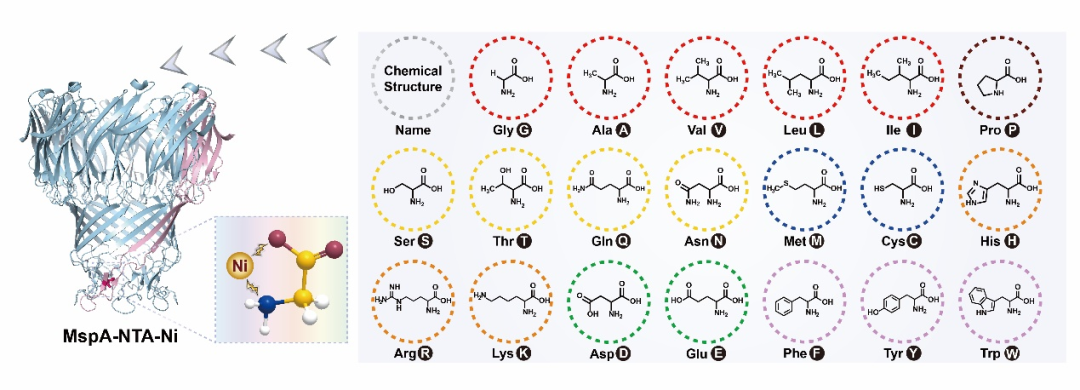
图:镍离子修饰纳米孔道实现20种蛋白质氨基酸全分辨
原文信息
Wang K, Zhang S, Zhou X, Yang X, Li X, Wang Y, et al. Unambiguous discrimination of all 20 proteinogenic amino acids and their modifications by nanopore. Nature Methods 2024;21:92–101. PMID: 37749214.
原文链接
https://doi.org/10.1038/s41592-023-02021-8

▲ 长按阅读原文