

ADMIN | 2025-03-28 01:12:40.0


人类跨脑区细胞图谱—Brain Cell Atlas
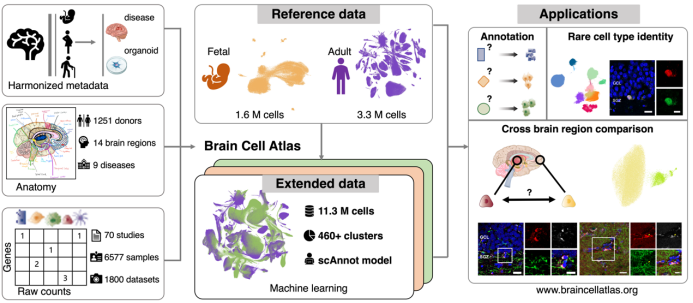
图:人类跨脑区细胞图谱—通过大规模数据整合发现稀有细胞类型
数据库链接
www.braincellatlas.org
原文信息
Chen X, Huang Y, Huang L, Huang Z, Hao ZZ, Xu L, et al. A brain cell atlas integrating single-cell transcriptomes across human brain regions. Nature Medicine 2024;30:2679–91. PMID: 39095595.
原文链接
https://www.nature.com/articles/s41591-024-03150-z

▲ 长按阅读原文
基于培养的人类肠道真菌基因组描述肠道真菌生物群落及其疾病关联性
大连医科大学王超、马骁驰团队牵头,联合中国科学院上海药物研究所果德安团队和法国农业科学研究院Francis Martin团队,大规模培养了12,453株人体肠道真菌,获得760个基因组(含69个新基因组),涉及48个科、206个物种,明确了人群中高丰度真菌和广泛流行的真菌物种。聚焦炎性肠病相关的特征性肠道真菌,发现三株真菌能够有效缓解多种因素诱导的小鼠溃疡性结肠炎,并明确法尼醇类活性代谢物和法尼醇X受体作用靶点。该研究丰富了对肠道真菌组的分类学、功能和代谢多样性的理解,为真菌生物技术领域的未来研究开辟新的视野。
该成果发表于Cell
推荐理由:利用培养组技术得到了迄今最大的人类肠道真菌参考基因组目录
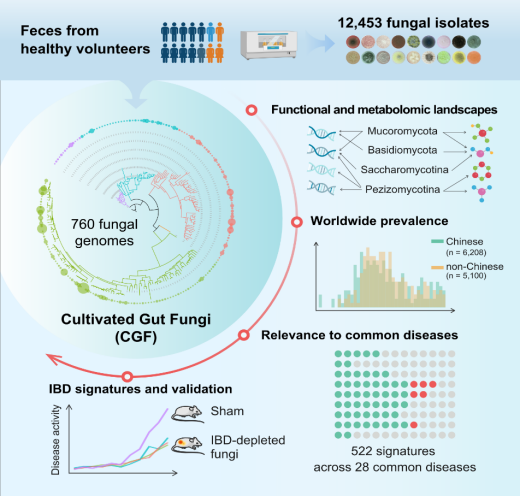
图:人肠道真菌组及疾病关联性分析
数据库链接
https://www.ncbi.nlm.nih.gov/bioproject/PRJNA833221
https://www.ncbi.nlm.nih.gov/bioproject/PRJNA83551
原文信息
Yan Q, Li S, Yan Q, Huo X, Wang C, Wang X, et al. A genomic compendium of cultivated human gut fungi characterizes the gut mycobiome and its relevance to common diseases. Cell 2024;187:2969–89.e24. PMID: 38776919.
原文链接
https://www.cell.com/cell/fulltext/S0092-8674(24)00469-0

▲ 长按阅读原文
AI2BMD实现量子级精度的蛋白质动力学模拟
分子动力学模拟是生命科学研究中通用的计算方法,已被广泛用于生物分子机理研究以及药物和疫苗的设计,而其方法的有效性和可靠性取决于其准确性和效率。微软研究院王童、邵斌团队提出了基于AI的量子级精度的生物分子动力学系统(AI2BMD),首次实现了量子级别精度的蛋白质动力学模拟。AI2BMD以量子级(从头计算)的精度高效地模拟了多种由超过1万个原子组成的全原子蛋白质,比量子力学方法快百万倍以上,为蛋白质折叠过程、构象空间探索、自由能等热力学性质的计算带来更为准确的描述和分析,从而为蛋白质动态机理探索、药物研发和蛋白质设计等领域带来全新的视角。
该成果发表于Nature
推荐理由:AI2BMD首次实现量子级别精度的蛋白质动力学模拟
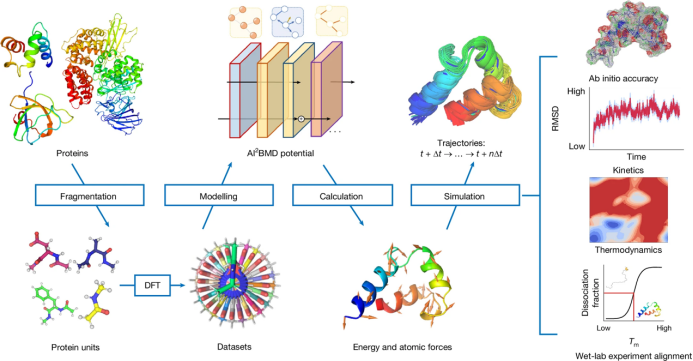
图:AI2BMD动力学模拟流程图
AI2BMD数据集、模型和模拟代码链接
https://github.com/microsoft/AI2BMD
原文信息
Wang T, He X, Li M, Li Y, Bi R, Wang Y, et al. Ab initio characterization of protein molecular dynamics with AI2BMD. Nature 2024;635:1019–27. PMID: 39506110.
原文链接
https://doi.org/10.1038/s41586-024-08127-z

▲ 长按阅读原文
鉴定同源长非编码RNA的新方法—lncHOME
该成果发表于Nature Genetics
推荐理由:开发的lncHOME方法可鉴定不同物种间的同源长非编码RNA

图:鉴定不同物种之间同源lncRNA方法(lncHOME)的计算流程
工具链接
https://github.com/huangwenze/lncHOME_analysis
原文信息
Huang W, Xiong T, Zhao Y, Heng J, Han G, Wang P, et al. Computational prediction and experimental validation identify functionally conserved lncRNAs from zebrafish to human. Nature Genetics 2024; 56:124–35. PMID: 38195860.
原文链接
https://doi.org/10.1038/s41588-023-01620-7

▲ 长按阅读原文
基于序列图像化策略的基因组结构变异检测和多样本分型—SVision-pro
该成果发表于Nature Biotechnology
推荐理由:基于“序列-图像”转换策略的SVision-pro创新算法支撑大规模专病队列和临床诊断数据中关键致病结构变异的发现

图:SVision-pro方法的序列图像化模块和神经网络识别模块
工具链接
https://github.com/xjtu-omics/SVision-pro
原文信息
Wang S, Lin J, Jia P, Xu T, Li X, Liu Y, et al. De novo and somatic structural variant discovery with SVision-pro. Nature Biotechnology 2025;43:181–5. PMID: 38519720.
原文链接
https://doi.org/10.1038/s41587-024-02190-7

▲ 长按阅读原文
基因序列数据库—GenBase
该成果发表于Genomics, Proteomics & Bioinformatics
推荐理由:国家生物信息中心核心数据库之一,对标GenBank,立足中国、服务全球

图:GenBase—规范、标准、智能、用户友好的基因序列数据汇交资源平台
数据库链接
https://ngdc.cncb.ac.cn/genbase/
原文信息
Bu C, Zheng X, Zhao X, Xu T, Bai X, Jia Y, et al. GenBase: a nucleotide sequence database. Genomics, Proteomics & Bioinformatics 2024;22:qzae047. PMID: 38913867.
原文链接
https://doi.org/10.1093/gpbjnl/qzae047

▲ 长按阅读原文
单细胞大规模基础模型—scFoundation
该成果发表于Nature Methods
推荐理由:全球首个参数规模超亿的单细胞转录组预训练模型scFoundation极大推动了AI驱动的科学研究范式转变
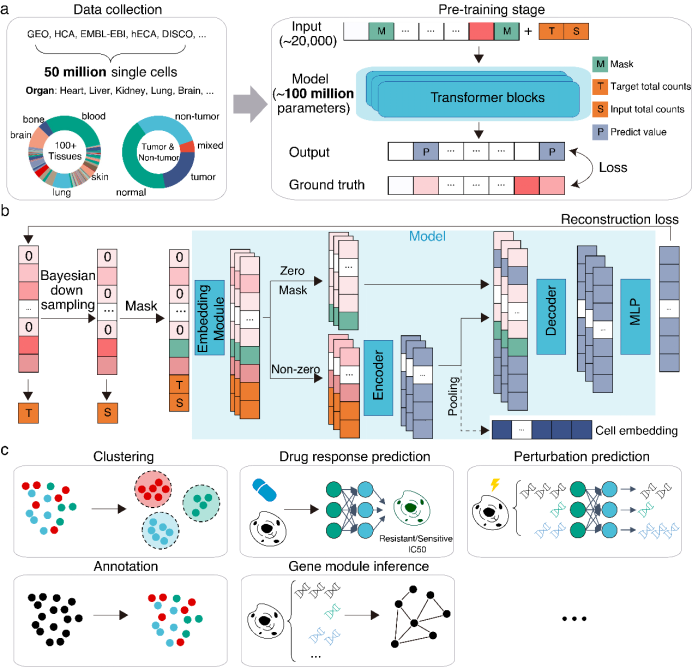
图:scFoundation预训练原理及其下游应用场景
工具链接
https://github.com/biomap-research/scFoundation
原文信息
Hao M, Gong J, Zeng X, Liu C, Guo Y, Cheng X, et al. Large-scale foundation model on single-cell transcriptomics. Nature Methods 2024;21:1481–91. PMID: 38844628.
原文链接
https://www.nature.com/articles/s41592-024-02305-7

▲ 长按阅读原文
泛癌种B细胞异质性的生物信息学解析
肿瘤浸润B细胞是多种癌症类型预后以及免疫治疗响应的标志物,但其在不同癌症类型中的异质性尚未得到系统性研究。北京大学张泽民、王东方团队与深圳湾实验室陈敏敏团队基于多癌种单细胞测序数据的系统性生物信息学整合,构建肿瘤原位B细胞成熟和激活的全景图谱,鉴定患者预后相关的关键B细胞亚型。研究发现稀有B细胞亚群肿瘤相关非典型B细胞(TAAB)和CD4 T细胞的互惠激活,揭示了肿瘤微环境免疫调控的新规律,挖掘B细胞作为预后、治疗响应新靶点的潜力。该研究从泛癌种视角为B细胞的异质性及其抗肿瘤免疫反应提供了新的见解,并为未来进一步探索B细胞在癌症中功能的共性和多样性奠定了基础。
该成果发表于Cell
推荐理由:构建高质量肿瘤浸润B细胞的泛癌单细胞图谱,为B细胞的异质性及其抗肿瘤免疫反应提供了新的见解

图:生物信息学整合分析揭示表型各异的肿瘤浸润B细胞亚类
数据库链接
http://pan-b.cancer-pku.cn/
原文信息
Yang Y, Chen X, Pan J, Ning H, Zhang Y, Bo Y, et al. Pan-cancer single-cell dissection reveals phenotypically distinct B cell subtypes. Cell 2024;187:4790–4811.e22. PMID: 39047727.
原文链接
https://doi.org/10.1016/j.cell.2024.06.038

▲ 长按阅读原文
哺乳动物高分辨率谱系追踪揭示肿瘤起源与进化新机制
哺乳动物体内细胞谱系复杂难以精确追踪。这极大限制了我们对肿瘤起源和进化机制的理解。中国科学院深圳先进技术研究院胡政团队与中山大学贺雄雷团队、何真团队合作,基于进化合成生物学策略首次建立了哺乳动物细胞高分辨谱系追踪技术和算法,重构了小鼠肠癌多阶段的高精度单细胞谱系树,揭示肠癌在初期是多克隆起源,随后转变为单克隆的进化发展模式,并解析了肿瘤从“温和”走向“凶恶”的关键基因和细胞互作机制。该研究突破了肿瘤是单克隆起源的传统认知,为理解肿瘤发生机制提供全新理论框架,有望推动肿瘤精准早筛和靶向干预的发展。
该成果发表于Nature
推荐理由:建立了哺乳动物细胞高分辨谱系追踪技术和算法,突破了经典的肿瘤单克隆起源理论,首次提出从多克隆到单克隆转变的早期肿瘤演化模式
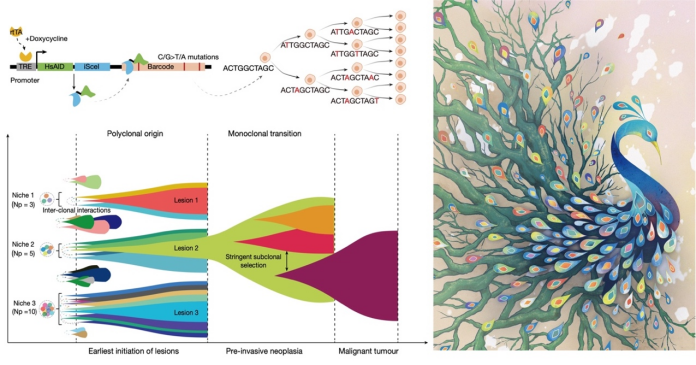
图:高分辨率谱系追踪揭示肿瘤从多克隆至单克隆转换的进化发展模式
数据库链接
https://github.com/zhaolianlu/SMALT-mouse
https://github.com/zhaolianlu/Homo-preCRC
https://smalt-phylogeny.org
原文信息
Lu Z, Mo S, Xie D, Zhai X, Deng S, Zhou K, et al. Polyclonal-to-monoclonal transition in colorectal precancerous evolution. Nature 2024;636:233–40. PMID:39478225.
原文链接
https://www.nature.com/articles/s41586-024-08133-1

▲ 长按阅读原文
AI重新定义病毒圈
病毒多样性的认知受限于基于同源性的发现方法,大量病毒仍处于未知状态。高效、准确地发现新病毒仍是挑战。中山大学施莽团队,阿里云李兆融团队与悉尼大学Edward Holmes团队共同研发了一种基于蛋白质序列和结构信息的深度学习语言模型来识别RNA病毒关键蛋白,准确率超过99%。利用该模型分析了10,487份宏转录组数据,发现超51万个RNA病毒基因组(contigs),涵盖16万余种RNA病毒及180个超群。其中23个超群无法通过同源性分析识别,被称为病毒圈的“暗物质”。这项研究将AI与病毒基因组发现结合,突破了传统的病毒发现方法的局限,扩展了人们对病毒圈的认知,率先将AI应用于病毒学领域,为未来AI在病毒学乃至更广泛的生命科学领域发挥核心作用奠定基础。
该成果发表于Cell
推荐理由:利用人工智能技术发现了180个病毒超群和16万余种全新RNA病毒,将已知病毒种类扩充了近30倍

图:AI识别潜在RNA病毒
工具链接
.https://github.com/alibaba/LucaProt
原文信息
Hou X, He Y, Fang P, Mei SQ, Xu Z, Wu WC, et al. Using artificial intelligence to document the hidden RNA virosphere. Cell 2024;187:6929–42.e16. PMID: 39389057.
原文链接
https://doi.org/10.1016/j.cell.2024.09.027

▲ 长按阅读原文