

ADMIN | 2021-03-08 10:29:35.0
为推动我国生物信息学的学科发展和创新研究,充分展示和宣传我国生物信息学领域的重大研究成果,《基因组蛋白质组与生物信息学报》(Genomics, Proteomics & Bioinformatics, 简称GPB)组织评选了2018年度“中国生物信息学十大进展”和2019年度“中国生物信息学十大进展”。在此基础上,GPB继续组织2020年度评选活动,经过推荐、初选和复选程序,现公布2020年度“中国生物信息学十大进展”(排名不分先后,按题目首字母顺序排序)。
感谢所有专家秉持专业和公正的态度参与本年度十大进展的推荐和评选,感谢我所NGDC冯昶瑞同学在本次活动资料整理和图片设计工作中所付出的努力!
祝贺所有入选工作的团队!GPB也祝愿大家在新的一年里身体健康、工作取得更多进展!
GPB
2021年3月8日

环形RNA精准定量和可变剪接体转换识别算法—CIRIquant
环形RNA是一类在真核生物中广泛存在的具有特殊环状结构的非编码RNA分子。能否从海量的测序数据中高效识别、组装和注释环形RNA及其不同形式的转录本,成为后续功能验证及表达调控机制研究的重要前提。中国科学院北京生命科学研究院赵方庆团队通过重构具有反向剪接特征的环形RNA参考序列,简化复杂的反向剪接位点比对问题,并结合测序读段比对到参考基因组和环形序列的结果,筛选出了高置信度的来自环形RNA的读段,解决了目前环形RNA识别和定量方法中准确度低和假阳性率高的问题。团队发现了两类环形RNA剪接体转换事件:线性/环形比例转换和成环位点使用偏好转换。该研究实现了对环形RNA及其母本基因的精确定量,为后续挖掘和筛选具有潜在功能的环形RNA提供了重要工具。
该成果发表于Nature Communications
工具链接:
https://sourceforge.net/projects/ciri
原文信息:
Jinyang Zhang, Shuai Chen, Jingwen Yang, Fangqing Zhao. Accurate quantification of circular RNAs identifies extensive circular isoform switching events. Nat Commun 2020; 11(1): 90.
原文链接:
https://www.nature.com/articles/s41467-019-13840-9
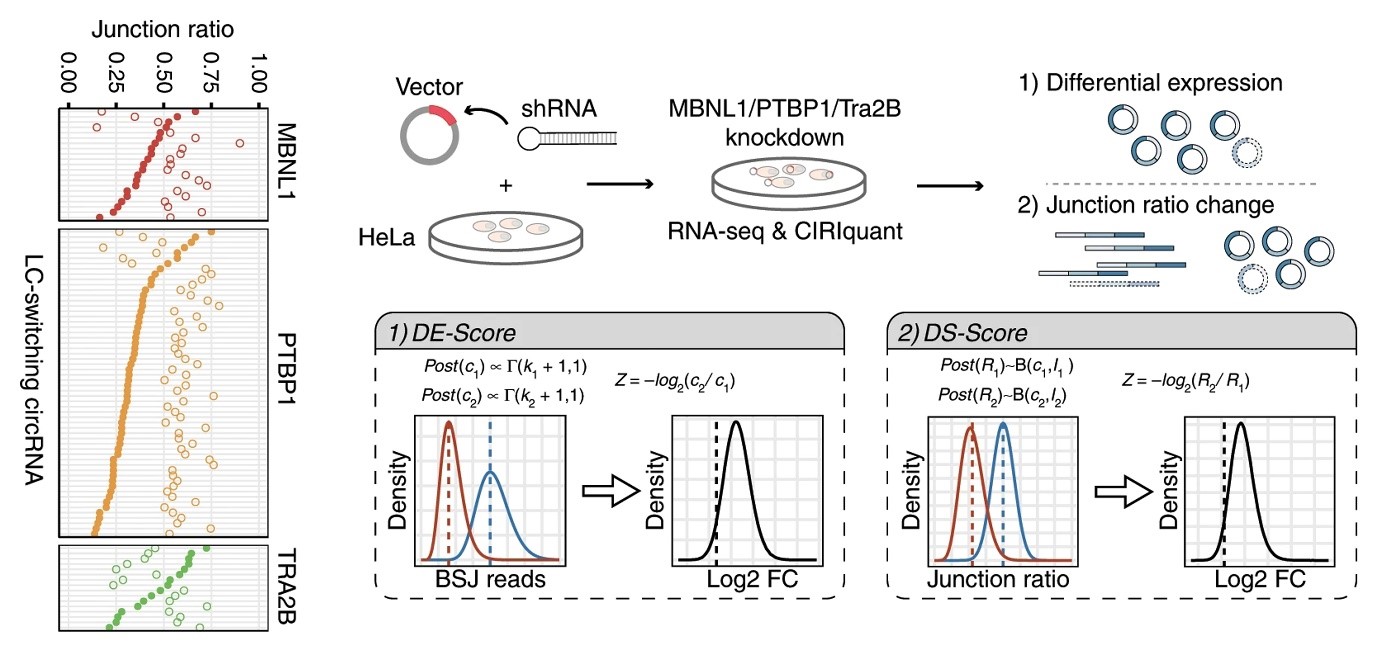
图:环形RNA的精确定量及剪接体转换的识别
环形RNA剪接过程中的两类环形RNA剪接体转换事件:线性/环形比例转换和成环位点使用偏好转换。
利用单细胞测序技术构建人类细胞图谱
长久以来,人体不同组织来源细胞的分析和鉴定,缺乏统一的标准,近几年涌现的单细胞测序技术有望建立一套系统性的细胞认知体系。浙江大学郭国骥团队通过高通量单细胞测序技术,分析了来自胎儿和成人共60种组织样品的70多万个细胞,绘制了全球第一张人类细胞图谱,首次从单细胞水平全面分析了涵盖八大系统的人体细胞种类,并揭示了一个普适性的哺乳动物细胞命运决定机制:细胞分化经历了一个从混乱到有序的发展过程。这项工作是人类细胞图谱计划的重要阶段性成果,描绘了人体细胞地图的基本框架,并定义了炎症上皮、炎症内皮及炎症基质等新细胞类型。人类细胞图谱计划将通过细胞数字化,极大地推动生物医学领域的基础与临床研究。
该成果发表于Nature
工具链接:
原文信息:
Xiaoping Han, Ziming Zhou, Lijiang Fei, Huiyu Sun, Renying Wang, Yao Chen, et al. Construction of a human cell landscape at single-cell level. Nature 2020; 581(7808): 303–9.
原文链接:
https://doi.org/10.1038/s41586-020-2157-4

图:涵盖人体各类组织细胞的首版人类细胞图谱
国家基因组科学数据中心建成生命健康多维数据资源体系
基因数据是国家重要基础性战略资源。针对我国基因组科学数据“存管用”的实际需求,解决数据安全、数据主权、数据孤岛等重大问题,中国科学院北京基因组研究所(国家生物信息中心)、上海营养与健康研究所和生物物理研究所在科技部、财政部支持下共同建设“国家基因组科学数据中心”(NGDC),面向我国人口健康、生物安全和重要战略生物资源,建立基因组科学数据汇交共享平台和多维组学数据资源系统,支撑我国科研项目数据的统一汇交和安全管理,形成基因组科学数据−信息−知识的多层次资源体系,为我国公益性科学研究与产业创新发展提供基础数据资源和共享平台。
该成果发表于Nucleic Acids Research
数据库链接:
原文信息:
National Genomics Data Center Members and Partners. Database resources of the National Genomics Data Center in 2020. Nucleic Acids Res 2020; 48(D1): D24–33.
原文链接:
https://doi.org/10.1093/nar/gkz913
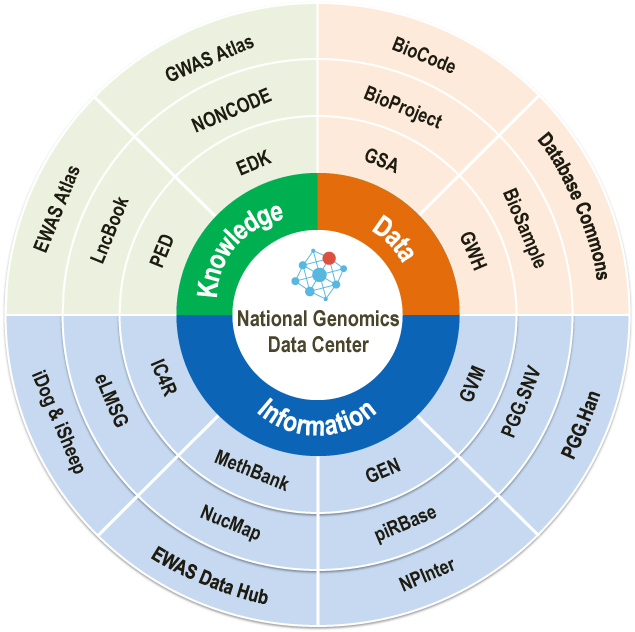
图:国家基因组科学数据中心核心数据资源
基于模糊布鲁因组装图的基因组组装算法—wtdbg2
如何快速准确地组装全基因组序列是第三代测序数据一直面临的难题。中国农业科学院农业基因组研究所阮珏与美国哈佛医学院李恒合作开发了基因组组装算法工具wtdbg2,将三代测序数据的组装效率提升4倍以上。在这项工作中,测序序列被切分为长度为256个碱基的单元块,进而以单元块为最小标识单位,构造了模糊布鲁因组装图。与经典的德布鲁因组装图相比,模糊布鲁因组装图具有摘要、稀疏等特性,充分发挥了序列长度在序列唯一性标识中的作用。基于模糊布鲁因组装图开发的算法工具wtdbg2可以直接处理未纠错的三代测序序列,并且在处理效率上处于领域前列。
该成果发表于Nature Methods
工具链接:
https://github.com/ruanjue/wtdbg2
原文信息:
Jue Ruan, Heng Li. Fast and accurate long-read assembly with wtdbg2. Nat Methods 2020; 17(2): 155–8.
原文链接:
https://www.nature.com/articles/s41592-019-0669-3

图:wtdbg2组装示意
每个小条块代表长度为256个碱基
基于人工智能度量学习的单细胞类型鉴定新方法—scLearn
随着海量的已标注细胞类型的单细胞转录组测序数据增多,利用这些数据作为参考数据集来鉴定新测序的细胞正成为一种细胞注释的新思路。现有的方法普遍存在两个不足:一是往往不能同时有效鉴定参考数据集中存在和缺失的细胞类型;二是鲁棒性不佳,无法适用于多个来源的数据集。同济大学刘琦团队基于人工智能度量学习,开发了单细胞类型鉴定新方法scLearn,提供了包含人类、小鼠在内的多个组织器官的预训练的完整参考数据集,对单标签以及多标签的细胞类型鉴定均适用,为有效利用海量的单细胞测序数据进行细胞类型鉴定提供了可借鉴的工具和资源。
该成果发表于Science Advances
工具链接:
https://github.com/bm2-lab/scLearn
原文信息:
Bin Duan, Chenyu Zhu, Guohui Chuai, Chen Tang, Xiaohan Chen, Shaoqi Chen, et al. Learning for single-cell assignment. Sci Adv 2020; 6(44): eabd0855.
原文链接:
https://doi.org/10.1126/sciadv.abd0855
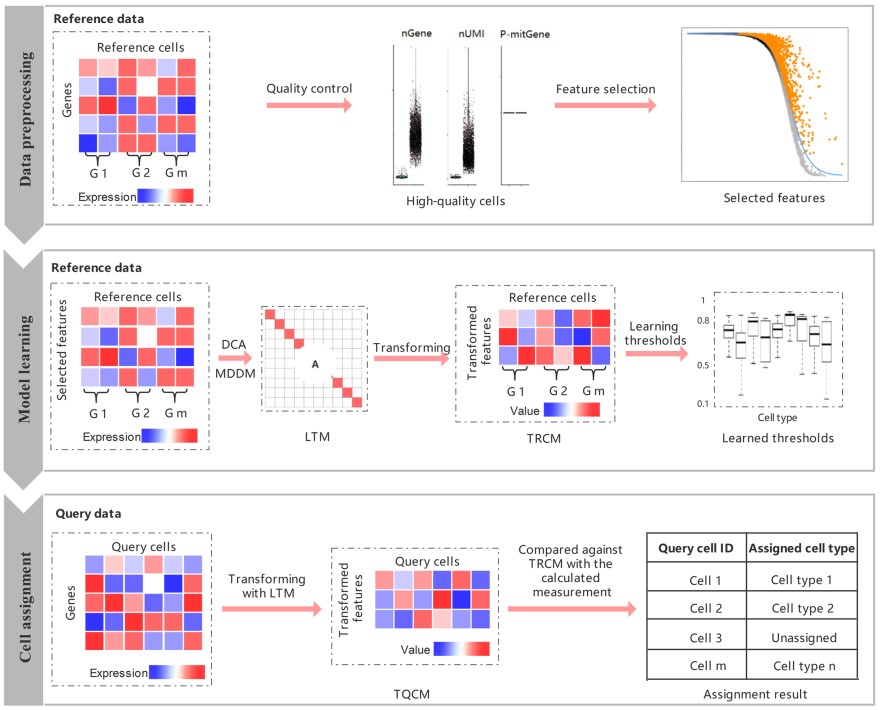
图:scLearn工作框架
实验验证的microRNA靶基因数据库—miRTarBase
MicroRNA(miRNA)是一类不编码蛋白质的遗传信息分子,在细胞分化、生物发育及疾病发生发展中发挥巨大作用。因此,揭示miRNA介导的遗传信息传递方式、靶基因和调控网络尤为重要。香港中文大学(深圳)黄宪达、李宗夷团队与深圳市龙岗区妇幼保健院魏凤香团队构建了迄今为止最完整的miRNA与靶基因交互作用(MicroRNA-Target Interaction,MTI)数据库(miRTarBase)。该数据库收录了提供实验证据支持MTI的文章共计超过10,000篇, 涉及来自32个物种的23,426个靶基因。随着CLIP-seq数据集的增加,目前miRTarBase 涵盖的MTI数量将近480,000个。通过自然语言技术搜集更全面的靶向关系及网络功能和注释信息,高度整合有用数据内容,并改善miRNA调控和存在的相关信息,miRTarBase为使用者提供更高效的体验。藉由miRNA调控靶基因信息与表现趋势,解析miRNA在调控特定生物代谢路径与不同癌症或复杂疾病的致病机制全貌。
该成果发表于Nucleic Acids Research
数据库链接:
http://mirtarbase.cuhk.edu.cn/
原文信息:
Hsi-Yuan Huang, Yang-Chi-Dung Lin, Jing Li, Kao-Yao Huang, Sirjana Shrestha, Hsiao-Chin Hong, et al. miRTarBase 2020: updates to the experimentally validated microRNA-target interaction database. Nucleic Acids Res 2020; 48(D1): D148–54.
原文链接:
https://doi.org/10.1093/nar/gkz896
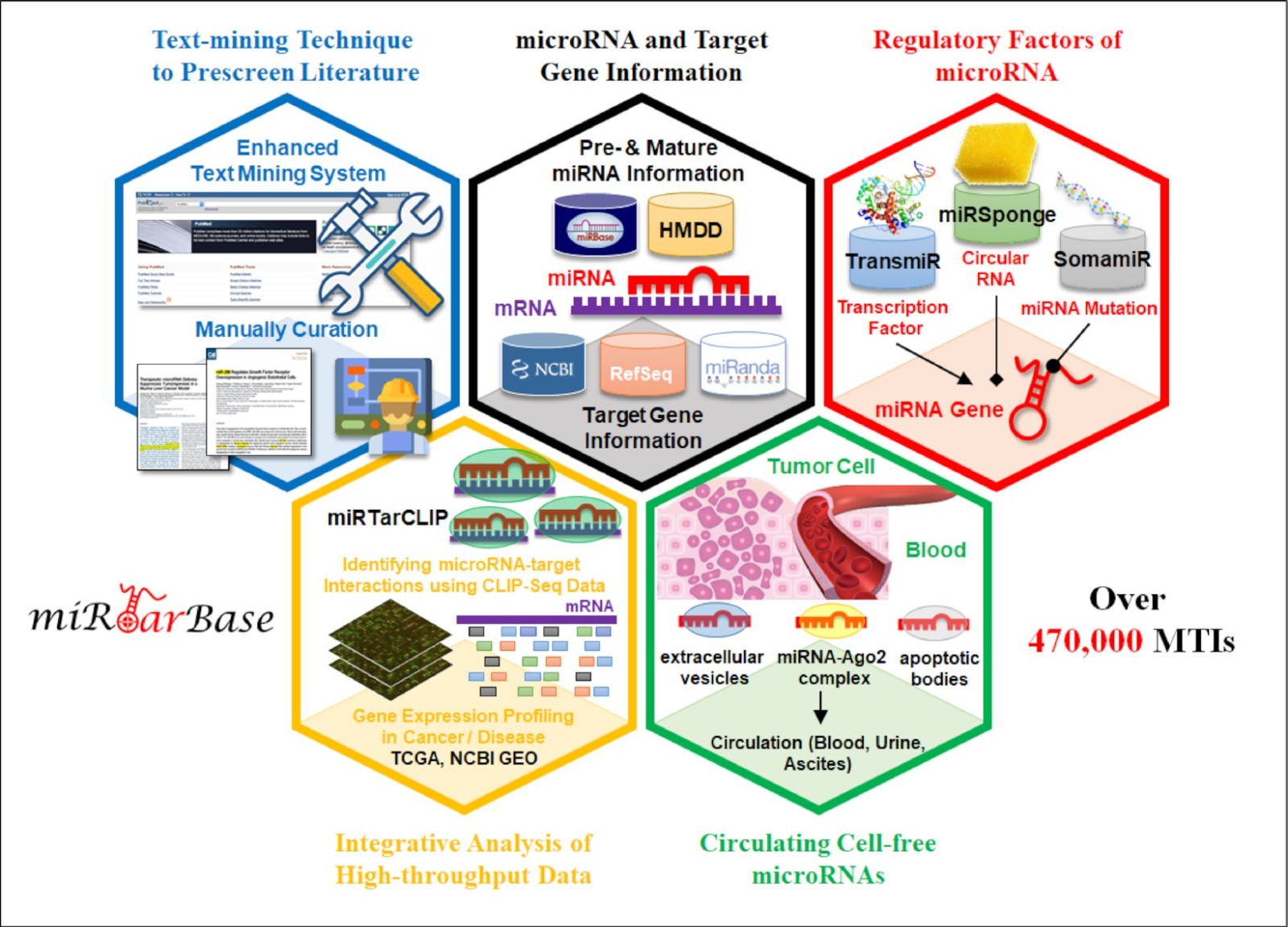
图:miRTarBase 2020 数据实现与亮点
miRTarBase 2020 通过整合自然语言处理技术、多组学实验数据与重要生物数据库,全面构建最完整的miRNA交互作用调控网络。
新冠肺炎临床信息综合数据库与人工智能诊断系统—iCTCF & HUST-19
新冠肺炎患者的临床数据整合和建模,能够为后续的临床决策提供重要的参考信息。华中科技大学王征、薛宇、王琳、史河水和曾玉兰团队,通过合作收集、整合和注释1500多例新冠肺炎患者的胸部CT影像和临床诊断大数据,构建了综合数据库iCTCF,数据容量为265.1 GB。在此基础上,团队设计了“基于混合学习的新冠无偏预测”人工智能诊断软件HUST-19,实现CT影像学和临床诊断数据的高效融合。该系统不仅可准确判断患者是否罹患新冠肺炎,还可以精确预测病情的严重程度以及潜在的死亡风险。iCTCF和HUST-19的相关数据和工具已无偿移交国家生物信息中心,有力地支持了我国抗疫相关的重大战略需求。
该成果发表于Nature Biomedical Engineering
数据库链接:
原文信息:
Wanshan Ning, Shijun Lei, Jingjing Yang, Yukun Cao, Peiran Jiang, Qianqian Yang, et al. Open resource of clinical data from patients with pneumonia for the prediction of COVID-19 outcomes via deep learning. Nat Biomed Eng 2020; 4(12): 1197–207.
原文链接:
https://www.nature.com/articles/s41551-020-00633-5
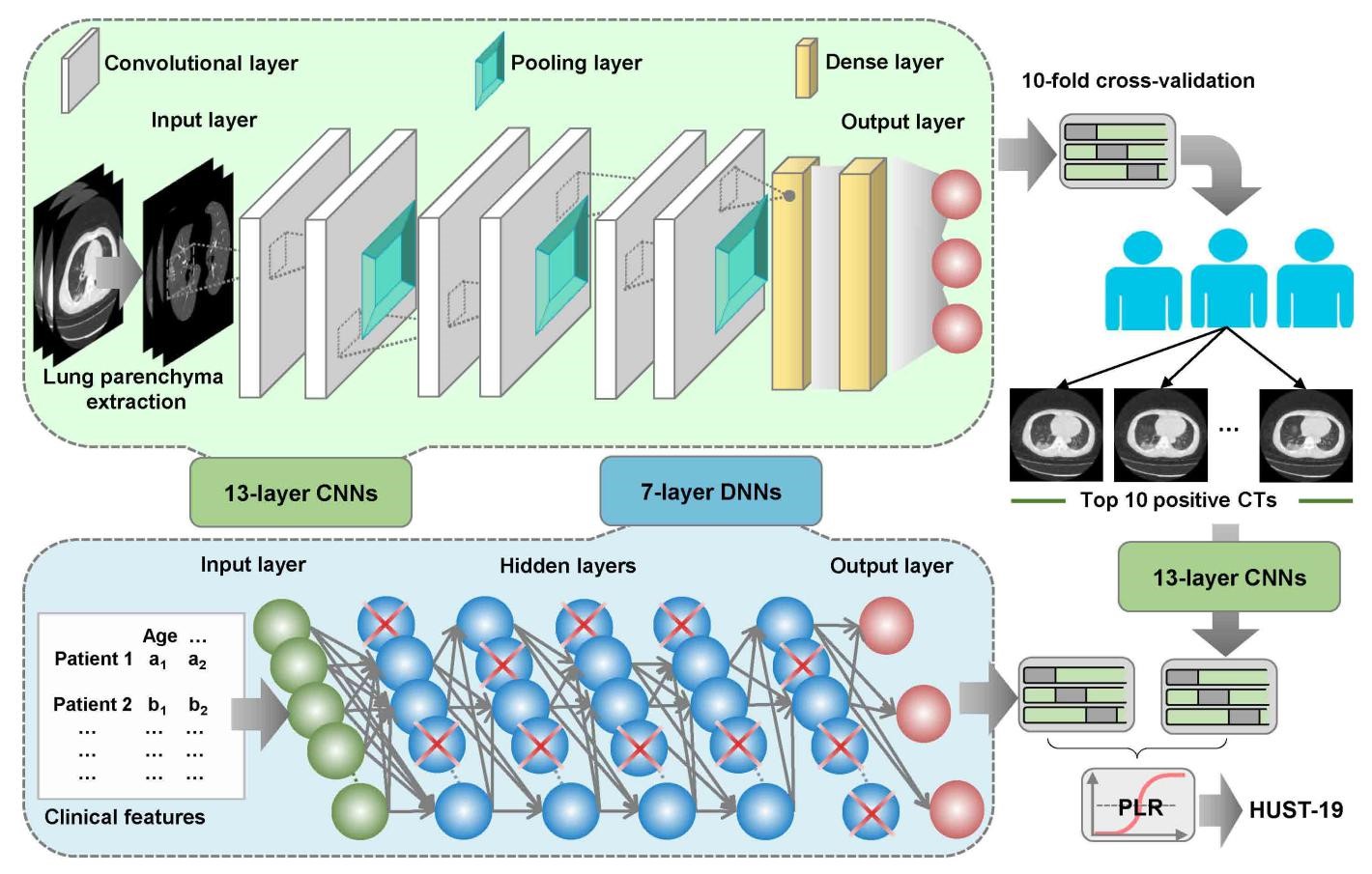
图: 基于混合学习的新冠无偏预测框架HUST-19
HUST-19包括一个用于预测单个CT切层的13层卷积神经网络框架,一个将基于单个切层预测转换为基于患者临床预后预测的13层卷积神经网络框架,一个利用新冠肺炎患者临床诊断信息预测患者临床预后的7层深度神经网络框架,和基于CT影像和临床诊断结果整合的惩罚逻辑回归算法,从而综合预测患者病情严重程度和死亡风险。
新冠病毒传播动力学模型—SAPHIRE
传染病模型研究对防控效果评估、揭示病毒传播特征、早期预警和防控政策制定具有重要价值。华中科技大学王超龙、邬堂春团队联合美国哈佛大学林希虹,基于经典的SEIR模型,结合病人出现症状前就具备传染性、大量感染者由于症状轻微而不易被发现等特征,并考虑人口流动和干预强度在不同阶段的变化,提出了命名为SAPHIRE的动力学模型,对武汉市新冠疫情流行曲线进行拟合分析,揭示了病毒的高传染性和高隐蔽性,并提示过早解除防控措施将大大增加疫情再次爆发的风险,为中国乃至全球的疫情防控政策制定提供了重要科学参考。
该成果发表于Nature
工具链接:
https://github.com/chaolongwang/SAPHIRE
原文信息:
Xingjie Hao, Shanshan Cheng, Degang Wu, Tangchun Wu, Xihong Lin, Chaolong Wang. Reconstruction of the full transmission dynamics of COVID-19 in Wuhan. Nature 2020; 584(7821): 420–4.
原文链接:
https://www.nature.com/articles/s41586-020-2554-8
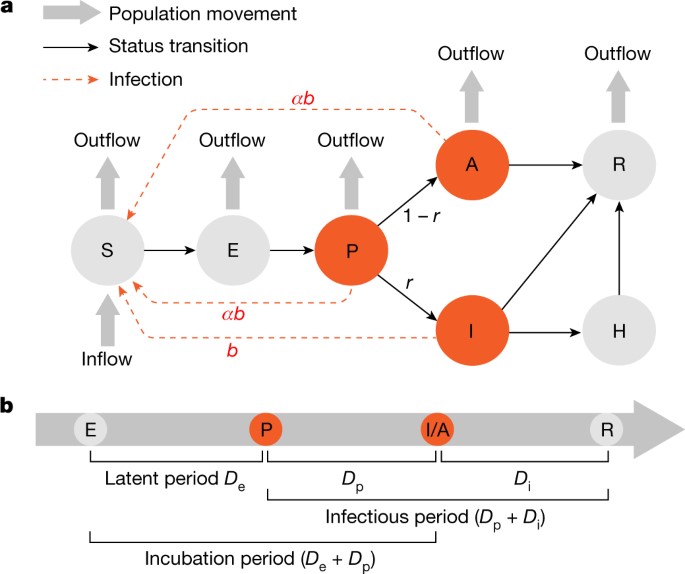
图:SAPHIRE动力学模型示意图
S代表易感人群,E代表潜伏期无传染性的感染者,P代表潜伏期有传染性的感染者,I为出现症状且被确诊的患者,A为未确诊患者,H为被隔离患者,R为康复或死亡人群。(a)SAPHIRE模型的不同人群(也称为“仓室”)之间的关系,其中b和r分别代表病毒的传染力和感染者的确诊率,是模型的主要参数,在不同阶段取值变化反映不同时间阶段综合干预措施的强度变化。(b)新冠感染者的病程及相关参数示意图。
单细胞转录组整合检索方法—Cell BLAST
作为细胞异质性研究的重要工具,近年来单细胞转录组测序技术蓬勃发展,产生了大量数据。为有效利用这些宝贵数据,北京大学高歌团队开发了单细胞转录组数据整合检索方法Cell BLAST。类比于生物序列研究中的BLAST算法,Cell BLAST可以准确快速地对新产生的单细胞数据在已有数据库中进行检索并注释,在节省了传统根据marker基因手动注释所需时间成本的同时,降低了人工操作可能引入的错误。Cell BLAST算法通过对抗学习有效地解决了单细胞转录组检索中复杂的多层次批次效应问题,并基于对单细胞测量过程内在随机性的刻画,提出了一个新的相似性度量指标NPD,可以有效应用于单细胞跨数据集整合检索和比较分析。为了充分发挥Cell BLAST的检索能力和作用,团队进一步自主构建了涵盖了多组织器官的跨物种单细胞转录组参考数据库ACA,并通过网页提供在线服务(https://cblast.gao-lab.org)。该工作为有效利用现有数据进行细胞注释和跨数据集研究提供了新的工具和资源,也展示了计算生物学、生物信息学方法在复杂生物学体系研究中的强大支撑作用。
该成果发表于Nature Communications
工具链接:
原文信息:
Zhi-Jie Cao, Lin Wei, Shen Lu, De-Chang Yang, Ge Gao. Searching large-scale scRNA-seq databases via unbiased cell embedding with Cell BLAST. Nat Commun 2020; 11(1): 3458.
原文链接:
https://doi.org/10.1038/s41467-020-17281-7
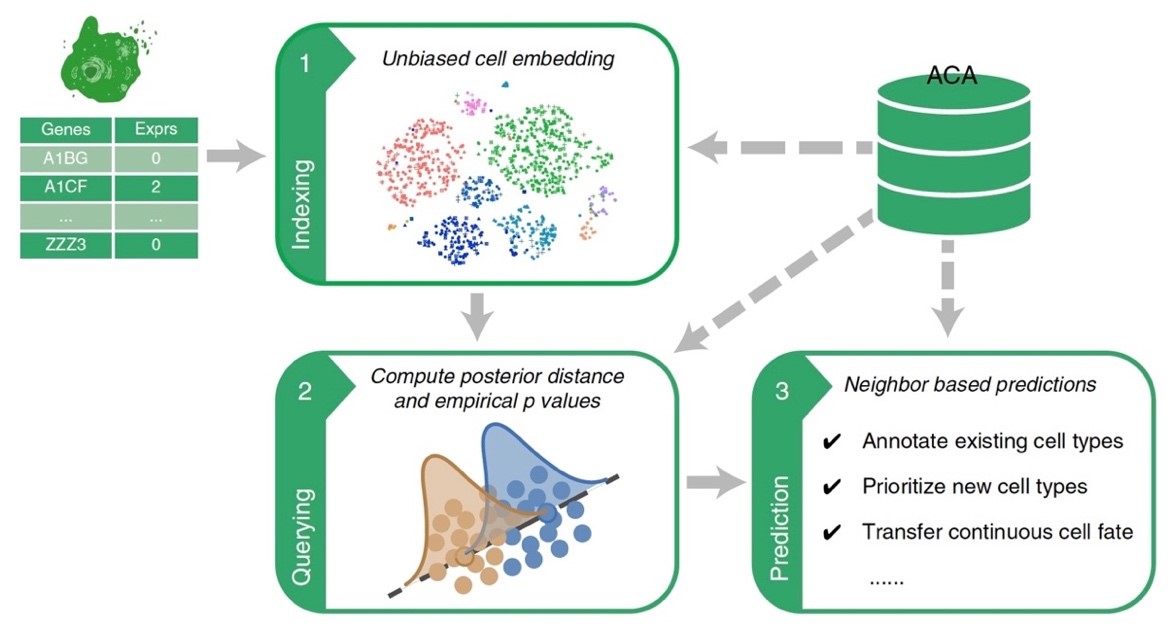
图:单细胞转录组检索方法Cell BLAST的工作流程
Cell BLAST首先将待查询数据与ACA数据库中的参考数据同时映射到低维细胞嵌入空间,在该空间中通过对抗学习消除多层次批次效应,并基于对单细胞测量内在随机性的刻画,使用NPD距离查询参考数据中与查询数据最相似的细胞,最后利用查询结果进行多角度注释。
2019新型冠状病毒信息库(2019nCoVR)及新冠病毒基因组、变异及单体型全景及演化
随着新冠病毒的全球大流行,新冠病毒基因组序列信息的实时整合与共享,序列变异的快速解析及动态演变监测有助于加速科学研究和知识发现,对医疗对策和防控决策的制定具有重要指导意义。中国科学院北京基因组研究所(国家生物信息中心)国家基因组科学数据中心新冠攻关团队实现了全球新冠病毒基因组数据信息的汇聚及质量评估,建立了新冠序列实时整合及变异时空动态变化的监测平台,构建了全球新冠病毒的单倍体型网络,实现了病毒演化谱系的实时追踪和变化监测,为新冠病毒的分子溯源、新冠疫情的科学防控提供了关键技术支撑与决策依据。
该成果发表于Genomics, Proteomics & Bioinformatics
数据库链接:
原文信息:
Shuhui Song, Lina Ma, Dong Zou, Dongmei Tian, Cuiping Li, Junwei Zhu, et al. The global landscape of SARS-CoV-2 genomes, variants, and haplotypes in 2019nCoVR. Genomics Proteomics Bioinformatics 2020.
原文链接:
https://doi.org/10.1016/j.gpb.2020.09.001
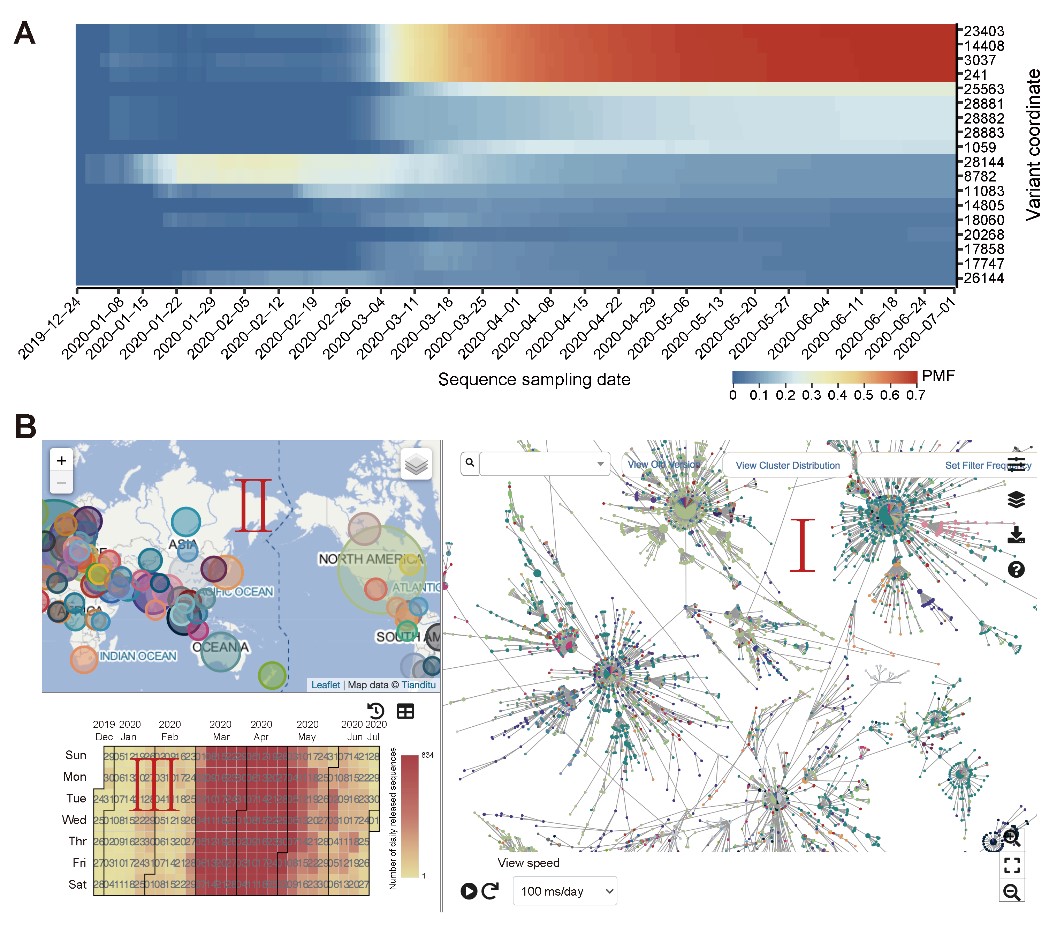
图:新冠病毒基因组序列变异及单体型演变监测
(A)序列突变群体发生率随时间的动态演变。(B)基因组单体型的时空演变。